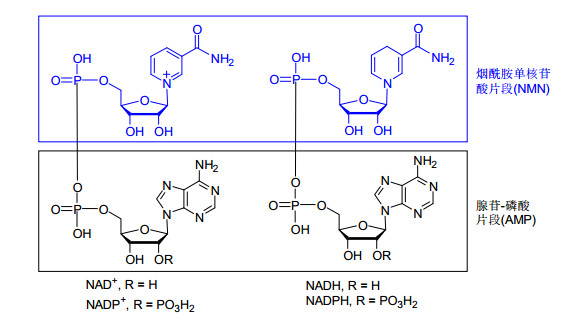
 图 1
吡啶核苷酸辅酶NAD+(NADH)的化学结构
Figure 1.
Chemical structure of pyridine nucleotide coenzyme NAD+(NADH)
图 1
吡啶核苷酸辅酶NAD+(NADH)的化学结构
Figure 1.
Chemical structure of pyridine nucleotide coenzyme NAD+(NADH)

Citation: Hou Shuhua, Qu Zhongguo, Zhong Keli, Bian Yanjiang, Tang Lijun. Recent Advances in Nicotinamide Adenine Dinucluotide (NAD+) Analogs Synthesis and Their Interactions with NAD+-Dependent Enzymes[J]. Chinese Journal of Organic Chemistry, 2015, 36(2): 297-305. doi: 10.6023/cjoc201508010

烟酰胺腺嘌呤二核苷酸(NAD+)类似物的合成及与NAD+依赖型酶相互作用进展
English
Recent Advances in Nicotinamide Adenine Dinucluotide (NAD+) Analogs Synthesis and Their Interactions with NAD+-Dependent Enzymes
-
Key words:
- NAD+
- / NAD+analog
- / NAD+-dependent enzyme
- / interaction
-
烟酰胺腺嘌呤二核苷酸(nicotinamide adenine dinucleotide, NAD+)以及它相应的还原型态(NADH), 就是我们通常所说的辅酶I, 是生物体内不可缺少的小分子化合物(结构如图 1)[1]. NAD+/NADH是电子载体, 在各种酶促氧化-还原反应中起着重要的作用.目前氧化还原酶中80%以NAD+/NADH为辅酶, 依赖于NAD+的脱氢酶至少催化6种不同类型的反应, 包括简单氢转移反应、脱氨基反应、氧化脱羧反应, 、醛氧化反应、碳-碳键氧化反应、碳-氮键氧化反应(表 1).除了氧化还原功能外, 在细胞的生长、分化、调节和疾病等非氧化还原生命过程中, NAD+同样起到非常重要的作用.如参与衰老相关的组蛋白脱乙酰化反应[2]、DNA修复过程中发挥重要作用的聚腺苷二磷酸核糖反应[3]、调控钙离子通道的腺苷二磷酸核糖环化反应[4]等. NAD+通常要和NAD+依赖型酶形成复合物后才能行使其生物化学功能, 可见它们之间复杂的相互作用对生命活动至关重要.同时由于NAD+依赖性酶利用共同的辅酶NAD+, 它们之间的非共价相互作用是NAD+依赖型酶催化机理研究中的共性和关键问题.通过研究NAD+与其依赖型酶相互作用, 有助于揭示酶与辅酶相互作用规律和化学本质.本文拟结合我们的工作将NAD+与其依赖型酶相互作用研究进展作一简要评述.

反应类型 还原态底物 氧化态产物 代表性酶 简单氢转移 

苹果酸脱氢酶乳酸脱氢酶乙醇脱氢酶 脱氨基反应 

谷氨酸脱氢酶 氧化脱羧反应 

异柠檬酸脱氢酶6-磷酸葡萄糖酸脱氢酶 醛氧化反应 

醛脱氢酶 碳-碳键氧化 

二氢类固醇脱氢酶 碳-氮键氧化 

二氢叶酸还原酶 表 1 典型的NAD+依赖型的氧化还原反应
Table 1. Typical redox reactions depending on NAD+1 NAD+类似物与天然酶相互作用
20世纪80年代前, 由于分析小分子与酶相互作用的各种先进手段还没有出现, 化学家们主要通过合成NAD+类似物来研究辅酶与酶相互作用规律. NAD+的化学改造及其类似物的功能研究, 始于20世纪50年代, 在20世纪70~90年代到达高峰.通过对NAD+分子的化学修饰及类似物的合成, 人们对NAD+与酶相互作用有一定的认识, 但由于当时对酶的结构认识比较有限, 对NAD+功能研究仅限于NAD+类似物的合成及类似物与天然酶相互作用规律.通过对NAD+及其类似物与NAD+依赖型酶相互作用早期工作的回顾, 有助于了解NAD+及其类似物研究的发展历程, 这将对合成新型NAD+类似物提供一定的指导.
1.1 NAD+分子的化学改造
NAD+是由烟酰胺单核苷酸(Nicotinamide mononucleotide, NMN)和腺苷一磷酸(Adenosine monophosphate, AMP)片段通过焦磷酸连接而成(图 1), 其中, NMN是其化学功能的主要承担者, 在氧化还原反应中起到传递电子和氢的作用, 而AMP部分则与功能蛋白质的有关结构域作用而锚定辅酶, 对辅酶的专一性识别和生化反应的立体选择性有重要影响.因此NAD+与其依赖型酶相互作用主要通过改变锚定酶的AMP部分来实现[5].
NAD+分子在生物体内的合成是极其复杂的, 不但需要消耗化学计量的原料, 同时消耗各种酶, 而有些酶在反应过程中必须要消耗等物质的量的辅因子才能使反应顺利进行, 需要的成本很高.同时由于酶具有很高的专一性, 不适用于合成与NAD+结构相差较大的类似物, 因此需要采用化学方法来合成NAD+类似物.
1.2 NAD+依赖型酶晶体结构
20世纪80年代结构生物学的兴起, 生物大分子的三维结构陆续得到阐明, 其中X射线晶体结构可以解析酶分子的三维结构, 有助于了解酶活性部位氨基酸残基所处的相对位置与实际状态, 以及与活性部位有关的其它基团[19].
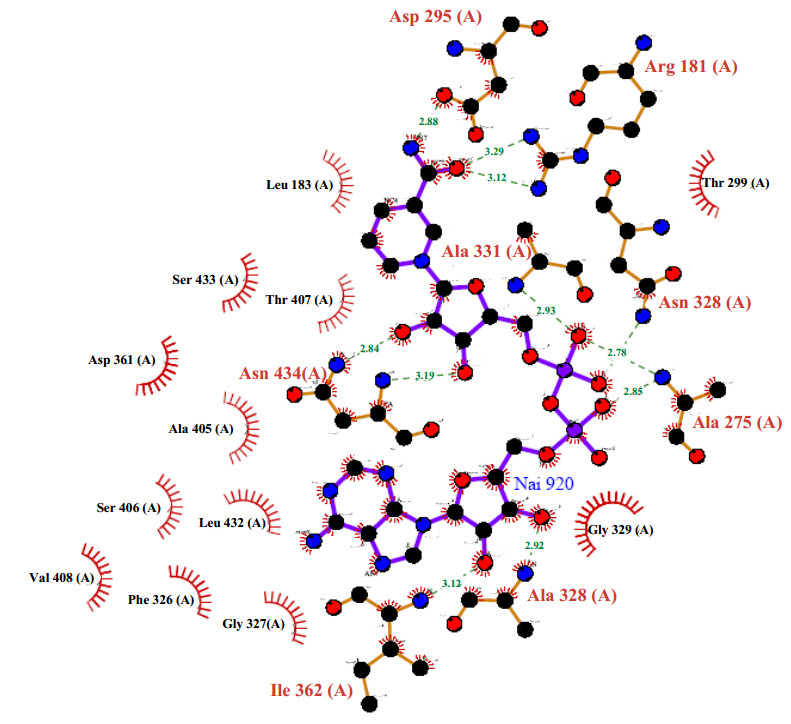
蛋白质与小分子的晶体结构为研究吡啶核苷酸辅酶及其依赖型脱氢酶相互作用提供了依据, 目前在蛋白质晶体结构数据库(Protein Data Bank, PDB)中能够搜索出2123种NAD+依赖型酶晶体结构, 并且数量在不断增加.根据酶晶体结构可以了解酶与NAD+相互作用的氨基酸残基, 进而阐明酶与辅酶相互作用规律[20].如在人线粒体中与NAD+中的AMP结合的具体氨基酸残基是K346、A312、G313、N259和E314[21], 猪蛔虫(1LLQ)中与NAD+中的AMP结合的具体氨基酸残基是L432、G327、I362、A328、G329、A330和N275[22](图 11).
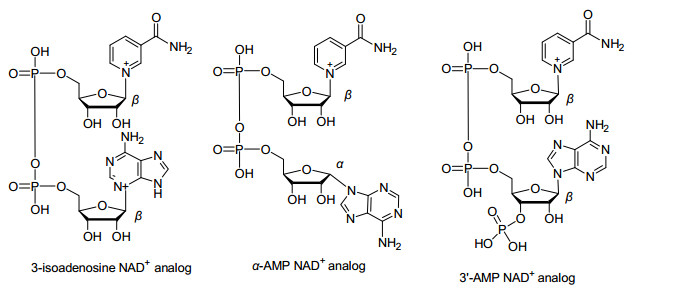
1.1.1 异构体的合成
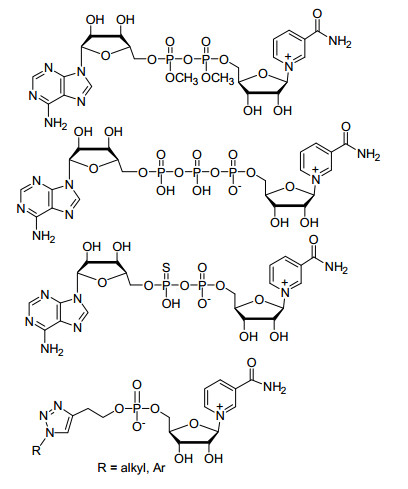
NAD(H)分子中腺嘌呤环含有多个N, 可以与核糖环形成糖苷键. Leonard教授合成了含3-isoadenosine的NAD+类似物, 其与不同脱氢酶相互作用活性与NAD+活性为6%~140%, NAD(H)分子中五元核糖均为β构型.为了考察核糖构型对酶活性的影响, Leonard等[6]合成了含α-AMP的NAD+类似物, 在不同的脱氢酶作用下, 可作为氢受体.为了考察磷酸对NAD(H)活性的影响, 合成了含3'-AMP的NAD+类似物, 其与脱氢酶作用完全没有活性(图 2).由此说明NAD(H)构型及NADP(H)中磷酸位置异构都会影响其与NAD(H)及NADP(H)依赖型酶相互作用.
1.1.4 糖环的改造
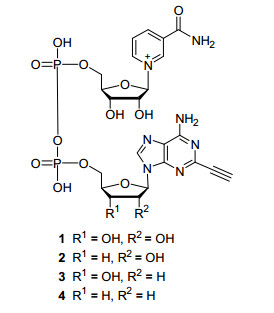
X-ray晶体结构表明, 呋喃糖环的2, 3位羟基会与脱氢酶的氨基酸残基相作用, 用脂肪链取代会导致类似物与酶作用的减弱, 这主要是由于类似物与酶形成复合物的过程受到了很大的空间位阻[15]. Marx等[16]合成4种改变腺苷核糖中羟基NAD+类似物, 并在腺嘌呤环2位上引入炔基以标记腺苷二磷酸核糖转移酶, 这4种NAD+类似物阐明了腺苷二磷酸核糖转移酶与NAD+相互作用关系, 证明了2位羟基在蛋白修饰中的重要性及腺苷二磷酸核糖转移酶的底物相容性(图 9).
1.1.5 焦磷酸的改造
通常焦磷酸键上的负电荷是影响功能位点氧化还原电位的因素之一, 改造焦磷酸键的类似物报道并不多见.通过焦磷酸的甲酯化、三磷酸化、用硫代磷酸等修饰, 这些类似物具有较高的氧化还原电位, 活性较低[17].将焦磷酸键变为单磷酸键的磷酸二酯型NAD+类似物对醇脱氢酶、乳酸脱氢酶均没有检测到活性[18](图 10).焦磷酸改造的NAD+类似物没有或者具有极低的辅酶活性, 说明焦磷酸键在NAD+与其依赖型酶相互作用中发挥着非常重要的作用.
至今报道的NAD+类似物中, 合成和分离难度较大, 合成周期较长.由于方法限制, NAD+类似物的改造研究只建立在NAD+结构的修饰及其与不同野生型NAD+依赖型酶的构效关系基础上, 化学和生物的信息量较少, 作用机制了解范围有限.
1.1.3 腺嘌呤环替换
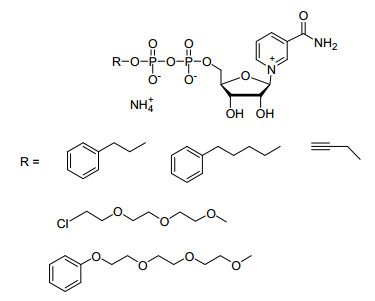
NAD(H)分子中AMP部分与功能蛋白质的有关结构域作用而锚定辅酶, 是NAD(H)与其依赖型酶相互作用的关键.因此, 我们课题组采用结构简单的脂肪链或芳香烃代替腺嘌呤环合成NAD+类似物, 与天然的苹果酸酶和乙醇脱氢酶相互作用, 具有一定的辅酶活性, 活性为NAD+的0.01%~30%[12], 说明此类结构简单的NAD+类似物与天然的苹果酸酶和乙醇脱氢酶有弱相互作用(图 6).我们[13]也利用点击化学合成含有三唑环的NAD+类似物, 此类NAD+类似物与天然的苹果酸酶和乙醇脱氢酶相互作用, 具有较低的辅酶活性.对天然的苹果酸酶, 含有三唑环的NAD+类似物的辅酶活性为NAD+的0.1%~4.5%;对天然的乙醇脱氢酶, 其辅酶活性为NAD+的0.2%~25.3%, 说明含有三唑环的NAD+类似物与天然的苹果酸酶和乙醇脱氢酶有弱相互作用, 合成的含有三唑环NAD+类似物含有不同官能团及不同分子体积, 利于解析酶与辅酶相互作用的规律(图 7).我们课题组[14]还利用胞嘧啶及其衍生物代替腺嘌呤环合成新型NAD+类似物, 此类NAD+类似物与天然苹果酸酶相互作用的kcat/Km为天然NAD+的0.09%~1.3%, 说明此类NAD+类似物与天然苹果酸酶间存在弱相互作用(图 8).
1.1.2 腺嘌呤环关键位点的修饰
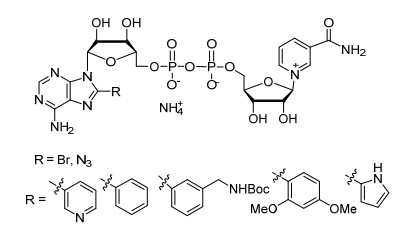
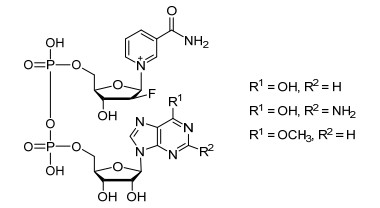
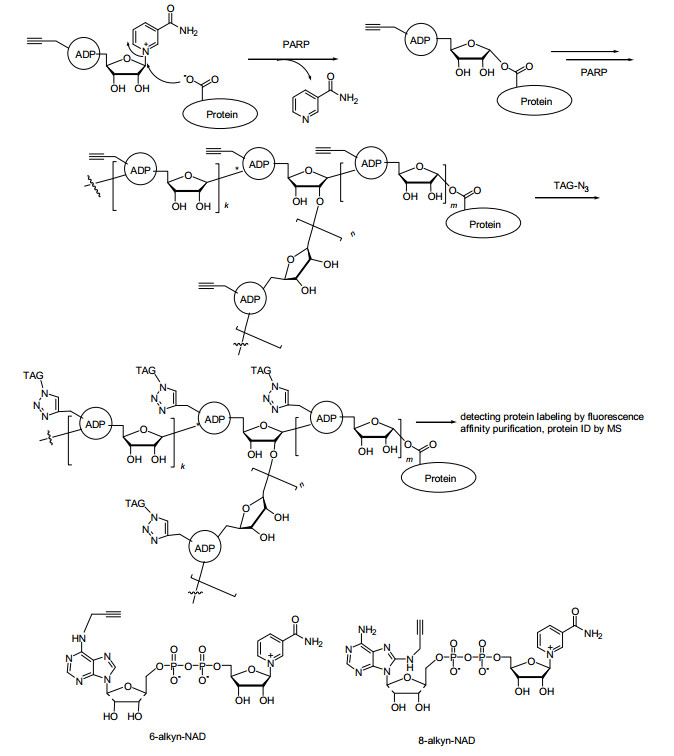
Windmüller和Kaplan[7]首先在富电子的腺嘌呤的N-1和N-6的位置进行烷基化并引入其他官能团, 得到了一系列的类似物.其中, 在N-1上引入其它基团, 辅酶活性大大降低, 而在N-6上引入其它官能团的NAD+类似物与NAD+依赖型脱氢酶的活性与NAD+与脱氢酶的活性相当. Koberstein[8]向嘌呤环的8位引入溴、叠氮基团, 实现了腺嘌呤环8位不同取代基NAD+类似物的合成, 此类化合物具有一定的酶活, 但是具有较高的km值, 说明与NAD+依赖型的脱氢酶相互作用减弱. Wagner等[9]在NAD+分子腺嘌呤环8位上修饰合成了NAD+荧光探针用于检测NAD+消耗型酶, 修饰之后的NAD+类似物仍然能够被NAD+依赖型酶识别, 说明两者之间存在相互作用(图 3).张礼和课题组[10]改变NAD+分子中腺嘌呤环2位和6位合成NAD+类似物, 发现这些类似物为腺苷二磷酸核糖环化酶CD38抑制剂, 利于解析NAD+类似物结构与活性相互关系(图 4). Lin等[11]在NAD+分子腺嘌呤环6位和8位上引入炔基合成新的NAD+类似物, 用来标记聚腺苷二磷酸核糖转移酶, 说明在腺嘌呤环的6位和8位进行修饰对NAD+与聚腺苷二磷酸核糖转移酶相互作用影响较小(图 5).
2 NAD+与突变酶相互作用
随着基因突变技术的发展, 生物化学家们通过基因突变的方式来研究小分子与生物大分子之间的相互作用.定点突变技术是对某个已知基因的特定碱基进行定点突变、缺失或者插入, 可以改变对应的氨基酸序列和蛋白质结构, 对突变基因的表达产物进行研究有助于了解蛋白质结构和功能的关系, 探讨蛋白质的结构/结构域[23]. Cook等[24]通过定点突变的技术确定了猪蛔虫中苹果酸酶D361与R370在与NAD+结合过程中的作用.通过定点突变改变酶利用辅酶能力也有很多报道, 比较成功的是改变辅酶的专一性, 如变NADP依赖型为NAD依赖型[25], 或者通过突变后, 利用辅酶的活性增加[26]. Muller-Steffner等[27]对NAD+关键活性位点定点突变, 通过NAD+与突变酶相互作用揭示了CD38/ NAD+糖水解酶催化机理.
3 NAD+类似物与突变酶相互作用
NAD+依赖性酶由于利用共同的辅酶, 辅酶浓度的变化会影响胞内所有NAD+依赖型酶活动, 进而引起代谢的广泛改变, 破坏细胞原有的生理状态, 对深入研究细胞内的氧化还原、糖代谢和能量代谢机制不利.因此, 在生物体内选择性、专一性研究或调控一种NAD+依赖型氧化还原酶而不干扰其它NAD+依赖型酶将有利于人们研究细胞内的代谢过程并对其进行调控.
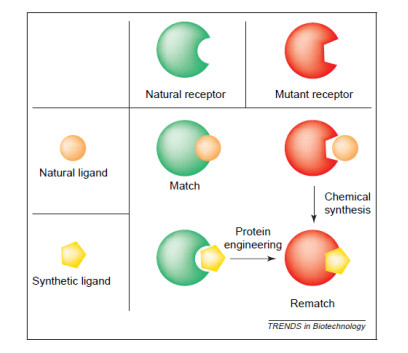
在生物体系中选择性或专一性研究或调控靶分子的功能非常困难, 常常会对其它生物大分子产生扰动, 必须合成新的高度特异性的蛋白质-配体对, 才能选择性地调节基因表达或细胞过程.生物正交体系的兴起为解决这一问题提供了可能[28].生物正交体系是一种将有机化学和分子生物学结合起来的化学生物学研究手段.它通过有机合成获得小分子化合物, 通过分子生物学段改造靶分子的高级结构, 在理想条件下, 合成小分子配体不能与未经改造的靶分子有效结合, 而与改造的靶分子很好的结合, 并表现出正常的活性; 而天然的小分子也不能与改造的靶分子有效结合(图 12)[29].这种新的高度特异性的蛋白质-配体对能够选择性地调控基因表达或细胞过程, 已引起了国内外广泛关注[30].
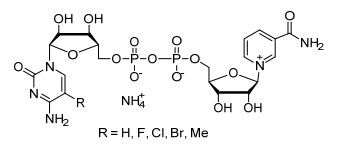
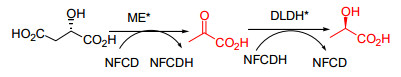
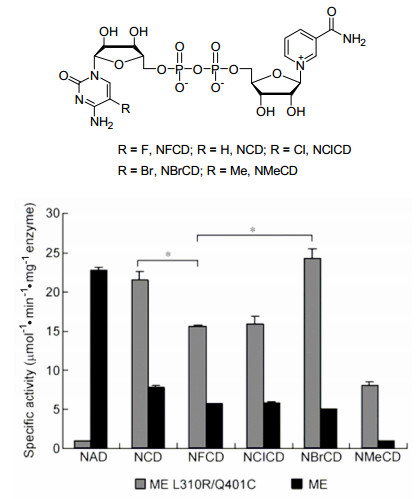
针对小分子配体与蛋白质相互作用的研究, 正交体系所需要的三个要素是:合成可供筛选的小分子配体文库, 构建突变型蛋白质的表达文库, 小分子配体与蛋白质相互作用的高通量筛选.我们课题组[31]前期合成了大量NAD+类似物.这些类似物保留了催化反应活性中心NMN部分, 改变了与酶结合的AMP部分, 包括简单脂肪烃、芳香烃、胞嘧啶及其衍生物、三唑环取代AMP的NAD+类似物.同时成功地构建了苹果酸酶突变库.通过与对辅酶结合域突变的苹果酸酶文库高通量筛选, 获得两种生物正交催化体系.一种是5-氟胞嘧啶二核苷酸(NFCD)与突变的苹果酸酶高通量筛选, 得到突变苹果酸酶ME-L310R/Q401C.这种突变酶对NFCD具有较高的催化活性, 而对NAD+具有很低的催化活性; 同时天然的苹果酸酶对NFCD具有很低的催化活性, 因此建立了一种新型的生物正交突变酶-辅酶配体, 可以用来选择性研究苹果酸酶.通过基因序列分析, 对D-乳酸脱氢酶(DLDH)和苹果酸脱氢酶(MDH)定点突变获得突变体DLDH-V152R和MDH-L6R, 能有效利用NFCD的还原态NFCDH, 实现了NFCD的循环再生, 使NFCD在催化转化L-苹果酸时只需要催化量[14a](图 13).同时, 也考察了胞嘧啶环上不同取代基对辅酶与野生型酶和突变酶相互作用关系[14b].胞嘧啶二核苷酸和5-溴胞嘧啶二核苷酸与突变苹果酸酶(ME L310R/Q401C)反应活性与野生型苹果酸酶与NAD+反应活性相当, 证明了NAD+类似物与突变酶功能配对可以代替野生型酶与NAD+配对行使其生化功能.同时取代基体积增大, 对突变酶和野生型酶选择性增强.当取代基为甲基时几乎不能够被野生型苹果酸酶识别, 而对突变酶ME L310R/Q401C具有较高的催化活性, 从而得到新型生物正交催化体系(图 14).
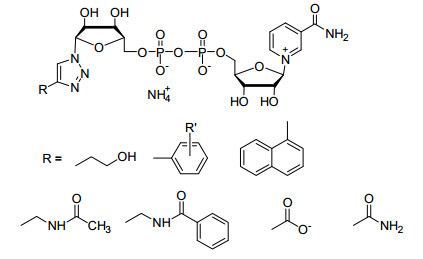
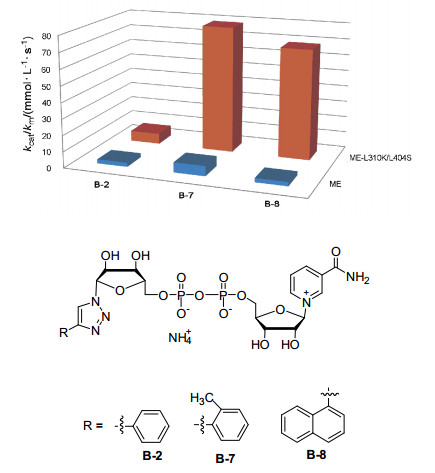
另外一种是含有三唑环的NAD+类似物与突变苹果酸酶文库筛选, 得到突变体ME-L310K/L404S.野生型苹果酸酶几乎不能利用NAD+类似物, 但是经过突变后获得的苹果酸酶ME-L310K/L404S可以较好地利用B-7和B-8, 而对天然NAD+分子具有很低的识别活性, 建立了一种新型的生物正交催化体系[32](图 15). B-2、B-7、B-8均为含有三唑环的NAD+类似物, 从三者的结构关系可以看出, 苯环上取代基大, 正交性增强.化合物B-2与突变苹果酸酶和野生型苹果酸酶相互作用力差别不大; 化合物B-7与化合物B-2相比增加一个甲基, 与突变酶的相互作用增强; 化合物B-8将化合物B-2中苯环变为萘环, 活性也增加, 体现出优异的生物正交性.
NAD+类似物与突变苹果酸酶生物正交体系的成功构建, 可以为其它NAD+依赖型酶(如乳酸脱氢酶、乙醇脱氢酶、谷氨酸脱氢酶等)正交体系的建立提供理论依据和技术支持.这两种生物正交催化体系的建立证明了NAD+类似物可以与NAD+依赖型酶突变库中的酶形成特异性配对, 这种高度特异性的NAD+类似物-NAD+依赖型酶突变体配对即NAD+依赖型生物正交氧化还原体系将有助于人们选择性和专一性研究一种NAD+依赖型酶, 研究其参与细胞活动的机理, 为研究细胞内的代谢过程及调控过程提供科学依据.
4 总结与展望
生命活动的正常进行离不开NAD(H)的参与, NAD+类似物的合成及与NAD+依赖型酶相互作用的持续研究和不断进展已经为探索酶与辅酶相互作用规律和化学本质提供了理论依据, 但是在细胞内选择性、专一性研究一种NAD+依赖型酶参与生命活动的机制仍然具有很大的挑战性.生物体内含有大量NAD+依赖性酶, 由于利用共同的辅酶, 辅酶浓度的变化会影响胞内所有NAD+依赖型酶活动, 对深入研究细胞生命活动不利.在生物体内选择性、专一性研究或调控一种NAD+依赖型氧化还原酶而不干扰其它NAD+依赖型酶将有利于人们研究细胞内的代谢过程并对其进行调控将成为本领域的热点前沿.目前我们已经在胞外建立了新型生物正交催化体系, 在不久的将来胞内生物正交体系的建立将大大增进人类对NAD+参与生命活动深入和崭新的理解.同时, 为在分子水平上选择性研究和调控辅酶依赖型生物大分子的生理生化功能提供化学生物学研究新方法, 为代谢流的调控提供新工具.
-
-
[1]
Imai, S. Biochim. Biophy. Acta 2010, 1804(8), 1584.
(b) Houtkooper, R. H.; Cantó, C.; Wanders, R. J.; Auwerx, J. Endocr. Rev. 2010, 31(2), 194.
(c) Xia, W. L.; Wang, Z.; Wang, Q.; Han, J.; Zhao, C. P.; Hong, Y. Y.; Zeng, L. L.; Tang, L.; Ying, W. H. Curr. Pharm. Des. 2009, 15(1), 12. -
[2]
Schmeisser, K.; Mansfeld, J.; Kuhlow, D.; Weimer, S.; Priebe, S.; Heiland, I.; Birringer, M.; Groth, M.; Segref, A.; Kanfi, Y.; Price, N. L.; Schmeisser, S.; Schuster, S.; Pfeiffer, A. F. H.; Guthke, R.; Platzer, M.; Hoppe, T.; Cohen, H. Y.; Zarse, K.; Sinclair, D. A.; Ristow, M. Nat. Chem. Biol. 2013, 9(11), 693.
(b) Imai, S.; Guarente, L. Trends Cell Biol. 2014, 24(8), 464.
(c) Dudev, T.; Lim, C. J. Am. Chem. Soc. 2010, 132(46), 16533. -
[3]
Tallis, M.; Morra, R.; Barkauskaite, E.; Ahel, I. Chromosoma 2014, 123(1~2), 79.
(b) Scobie, K. N.; Damez-Werno, D.; Sun, H. S.; Shao, N. Y.; Gancarz, A.; Panganiban, C. H.; Dias, C.; Koo, J.; Caiafa, P.; Kaufman, L.; Neve, R. L.; Dietz, D. M.; Shen, L.; Nestler, E. J. Proc. Natl. Acad. Sci. U. S. A. 2014, 111(5), 2005.
(c) Jiang, H.; Kim, J. H.; Frizzell, K. M.; Kraus, W. L.; Lin, H. J. Am. Chem. Soc. 2010, 132(27), 9363 -
[4]
Graeff, R.; Liu, Q.; Kriksunov, I. A.; Kotaka, M.; Oppenheimer, N.; Hao, Q.; Lee, H. C. J. Biol. Chem. 2009, 284(40), 27629. doi: 10.1074/jbc.M109.030965
-
[5]
Giangreco, I.; Packer, M. J. J. Med. Chem. 2013, 56(15), 6175. doi: 10.1021/jm400644z
-
[6]
Leonard, N. J.; Laursen, R. A. Biochemistry (Mosc.) 1965, 4(2), 365. doi: 10.1021/bi00878a028
-
[7]
Windműeller, H.; Kaplan, N. J. Biol. Chem. 1961, 236(10), 2716.
(b) Woenckhaus, C.; Koob, R.; Burkhard, A.; Schaefer, H. G. Bioorg. Chem. 1983, 12(1), 45. -
[8]
Koberstein, R. Eur. J. Biochem. 1976, 67(1), 223. doi: 10.1111/ejb.1976.67.issue-1
-
[9]
Pergolizzi, G.; Butt, J. N.; Bowater, R. P.; Wagner, G. K. Chem. Commun. 2011, 47(47), 12655. doi: 10.1039/c1cc15499k
-
[10]
Wang, S.; Zhu, W.; Wang, X.; Li, J.; Zhang, K.; Zhang, L.; Zhao, Y.-J.; Lee, H. C.; Zhang, L. Molecules 2014, 19(10), 15754. doi: 10.3390/molecules191015754
-
[11]
Jiang, H.; Kim, J. H.; Frizzell, K. M.; Kraus, W. L.; Lin, H. J. Am. Chem. Soc. 2010, 132(27), 9363. doi: 10.1021/ja101588r
-
[12]
侯淑华, 刘武军, 赵宗保, 有机化学2012, 32(2), 349. doi: 10.6023/cjoc1106032Hou, S. H.; Liu, W. J.; Zhao, Z. B. Chin. J. Org. Chem. 2012, 32, 349 (in Chinese). doi: 10.6023/cjoc1106032
-
[13]
Hou, S.; Liu, W.; Ji, D.; Wang, Q.; Zhao, Z. K. Tetrahedron Lett. 2011, 52(44), 5855. doi: 10.1016/j.tetlet.2011.08.152
-
[14]
Ji, D.; Wang, L.; Hou, S.; Liu, W.; Wang, J.; Wang, Q.; Zhao, Z. K. J. Am. Chem. Soc. 2011, 133(51), 20857.
(b) Ji, D.; Wang, L.; Liu, W.; Hou, S.; Zhao, K. Z. Sci. China Chem. 2013, 56(3), 296. -
[15]
Imai, T.; Okuda, S.; Suzuki, S. J. Biol. Chem. 1969, 244(17), 4547.
(b) Suhadolnik, R. J.; Lennon, M. B.; Uematsu, T.; Monahan, J. E.; Baur, R. J. Biol. Chem. 1977, 252(12), 4125. -
[16]
Wang, Y.; Roesner, D.; Grzywa, M.; Marx, A. Angew. Chem., Int. Ed. 2014, 53(31), 8159. doi: 10.1002/anie.201404431
-
[17]
Pfleiderer, G.; Sann, E.; Stock, A. Chem. Ber. 1960, 93(12), 3083. doi: 10.1002/(ISSN)1099-0682
-
[18]
Liu, W.; Wu, S.; Hou, S.; Zhao, Z. K. Tetrahedron 2009, 65(40), 8378. doi: 10.1016/j.tet.2009.08.007
-
[19]
Zhang, H. M.; Graeff, R.; Chen, Z.; Zhang, L. R.; Zhang, L. H.; Lee, H. C.; Hao, Q. A. J. Mol. Biol. 2011, 405(4), 1070. doi: 10.1016/j.jmb.2010.11.044
-
[20]
Egea, P. F.; Muller-Steffner, H.; Kuhn, I.; Cakir-Kiefer, C.; Oppenheimer, N. J.; Stroud, R. M.; Kellenberger, E.; Schuber, F. PLoS One 2012, 7(4), e34918. doi: 10.1371/journal.pone.0034918
-
[21]
Chang, G. G.; Tong, L. Biochemistry (Mosc.) 2003, 42(44), 12721. doi: 10.1021/bi035251+
-
[22]
Coleman, D. E.; Rao, G. S. J.; Goldsmith, E.; Cook, P. F.; Harris, B. G. Biochemistry (Mosc.) 2002, 41(22), 6928. doi: 10.1021/bi0255120
-
[23]
Mahmud, A.; Hattori, K.; Hongwen, C.; Kitamoto, N.; Suzuki, T.; Nakamura, K.; Takamizawa, K. Biotechnol. Lett. 2013, 35(5), 769. doi: 10.1007/s10529-013-1144-3
-
[24]
Aktas, D. F.; Cook, P. F. Biochim. Biophys. Acta 2008, 1784(12), 2059. doi: 10.1016/j.bbapap.2008.07.014
-
[25]
Peregrina, J. R.; Herguedas, B.; Hermoso, J. A.; Martinez-Julvez, M.; Medina, M. Biochemistry (Mosc.) 2009, 48(14), 3109. doi: 10.1021/bi802077c
-
[26]
Woodyer, R.; van der Donk, W. A.; Zhao, H. M. Biochemistry (Mosc.) 2003, 42(40), 11604. doi: 10.1021/bi035018b
-
[27]
Kuhn, I.; Kellenberger, E.; Cakir-Kiefer, C.; Muller-Steffner, H.; Schuber, F. Biochim. Biophy. Acta 2014, 1844(7), 1317. doi: 10.1016/j.bbapap.2014.03.014
-
[28]
Sletten, E. M.; Bertozzi, C. R. Acc. Chem. Res. 2011, 44(9), 666. doi: 10.1021/ar200148z
-
[29]
Chockalingam, K.; Zhao, H. Trends Biotechnol. 2005, 23(7), 333. doi: 10.1016/j.tibtech.2005.05.002
-
[30]
Miyazaki, Y.; Imoto, H.; Chen, L.-C.; Wandless, T. J. J. Am. Chem. Soc. 2012, 134(9), 3942.
(b) Zhang, Y.; Chen, P.; Yao, Z. J. Chin. Sci. Bull. 2013, 58, 2872 (in Chinese).
(张艳, 陈鹏, 姚祝军, 科学通报, 2013, 58, 2872.)
(c) Sletten, E. M.; Bertozzi, C. R. Angew. Chem., Int. Ed. 2009, 48(38), 6974. -
[31]
Wang, J.; Zhang, S.; Tan, H.; Zhao, Z. J. Microbiol. Methods 2007, 71(3), 225. doi: 10.1016/j.mimet.2007.09.001
-
[32]
Hou, S.; Ji, D.; Liu, W.; Wang, L.; Zhao, Z. K. Bioorg. Med. Chem. Lett. 2014, 24, 1307. doi: 10.1016/j.bmcl.2014.01.047
-
[1]
-
表 1 典型的NAD+依赖型的氧化还原反应
Table 1. Typical redox reactions depending on NAD+

反应类型 还原态底物 氧化态产物 代表性酶 简单氢转移 

苹果酸脱氢酶乳酸脱氢酶乙醇脱氢酶 脱氨基反应 

谷氨酸脱氢酶 氧化脱羧反应 

异柠檬酸脱氢酶6-磷酸葡萄糖酸脱氢酶 醛氧化反应 

醛脱氢酶 碳-碳键氧化 

二氢类固醇脱氢酶 碳-氮键氧化 

二氢叶酸还原酶 -

 扫一扫看文章
扫一扫看文章
计量
- PDF下载量: 0
- 文章访问数: 281
- HTML全文浏览量: 84


 下载:
下载:














 下载:
下载:

