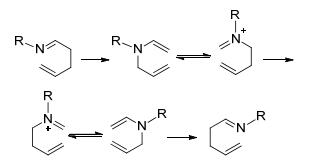
 图式1
Aza-cope重排的机理
图式1.
Mechanism of aza-cope reaction
图式1
Aza-cope重排的机理
图式1.
Mechanism of aza-cope reaction

Citation: Chen Yi, Hu Aohan, Yang Lingyi, Li Zaoying, Yan Kun. Recent Progress in Fluorescent Probe for the Detection of Reactive Carbonyl Species[J]. Chinese Journal of Organic Chemistry, 2017, 37(8): 1939-1951. doi: 10.6023/cjoc201703040

活性羰基化合物荧光探针的研究进展
English
Recent Progress in Fluorescent Probe for the Detection of Reactive Carbonyl Species
-
Key words:
- reactive carbonyl species
- / fluorescent probe
- / molecular design
- / formaldehyde
- / methylglyoxal
-
活性羰基化合物(Reactive carbonyl species, RCS)一直以来被人们称为致癌物和人类毒物, 它的来源分为两种:第一, 食物的加工过程; 第二, 生物体内碳水化合物、脂质以及氨基酸等物质氧化后的副产物[1].生物体内的活性羰基化合物是一类包含一个或多个羰基基团的高活性亲电化合物, 主要包括甲醛(FA)、甲基乙二醛(MGO)、丙二醛(MDA)、丙烯醛、丙醛、乙二醛、乙醛、4-羟基壬烯醛、草酰乙酸、丙酮酸等[2].研究表明, 正常情况下, 生物体内活性羰基化合物的浓度维持在一个正常的范围之内, 当其浓度升高时, 会导致多种疾病, 如各种癌症、神经紊乱、糖尿病以及心脏病等[3].考虑到生物体内羰基化合物的浓度与人体健康的重大关系, 探研一种高效的用来检测活性羰基化合物的技术显得至关重要.
传统的用来检测活性羰基化合物的方法主要包括4种, 即辐射测定法、气相色谱法、高效液相色谱法和比色法等[4].同样地, 它们能提供较高的灵敏性和选择性, 但是存在一些不足, 如样品处理较麻烦, 不能保证样品的完整性, 也不能用于生物体内RCS的实时探测.对于活性羰基化合物的检测, 传统的检测方法都存在一定的缺陷和不足, 而近期发展起来的荧光探针的检测方法常用于检测生物体内的一些物质, 如硫醇[5]、金属离子[6]、活性氧物种[7]以及活性氮物种[8]等, 具有操作简单、高灵敏性、高选择性、响应时间短而且检测过程对样品没有破坏, 对活细胞造成的伤害也较小, 能实现生物体内的实时监测等优点.至今为止, 荧光探针在生物体内的成像研究主要分为三个阶段, 即细胞内成像阶段[9], 活体组织成像阶段[10]以及动物体内活体成像阶段[11], 这三个阶段分布进行, 推进着荧光探针向不同领域的快速发展.目前荧光探针在生物体内的成像研究主要集中在活细胞成像阶段, 并且近年来相继报道一些靶向定位到某个特定的细胞器, 如线粒体[12]、溶酶体[13]和高尔基体[14]等的荧光探针分子, 引起了科研工作者的研究兴趣.
关于活性羰基化合物的荧光探针方面的研究一直是科研工作者的重点研究方向.关于这类物质在荧光探针方面的报道也不断增加, 但是, 关于活性羰基化合物荧光探针研究进展方面的综述甚少.本文通过探针分子与待测物之间发生的化学反应的类型, 总结了常见的活性羰基化合物如甲醛(FA)、甲基乙二醛(MGO)、丙二醛(MDA)、丙烯醛等物质的荧光探针, 并且对一些荧光探针的设计理念、识别机理以及应用进行了总结和归纳.
1 检测甲醛(FA)的荧光探针
甲醛, 作为结构最简单的醛类, 是一种具有高度反应活性的羰基物种.生物体主要通过脱甲基酶和氧化酶在体内产生甲醛[15], 当生物体内甲醛的浓度发生异常时, 会引发癌症[16]、神经性疾病[17]、糖尿病、肝病以及心脏病[18]等, 因此, 设计合成用于快速、高效、实时检测生物体内甲醛的探针, 在探究甲醛关于生理、病理的研究以及疾病诊断和控制等方面的应用意义重大.目前, 关于甲醛荧光探针的设计主要采用的反应机理有Aza-cope重排、NHNH2以及NH2与甲醛的反应、金属配合物与甲醛的反应, 以及其它反应类型五大类.
1.1 基于探针与甲醛发生Aza-cope反应的探针
Aza-cope重排反应作为有机化学中合成复杂有机化合物的重要方法, 它的重排机理如Scheme 1所示, 含氮的1, 5-二烯烃会发生cope重排一样的[3, 3]-键重排, 重排之后连续进行Mannich反应, 这种连续的反应被称为Aza-cope重排反应[19].
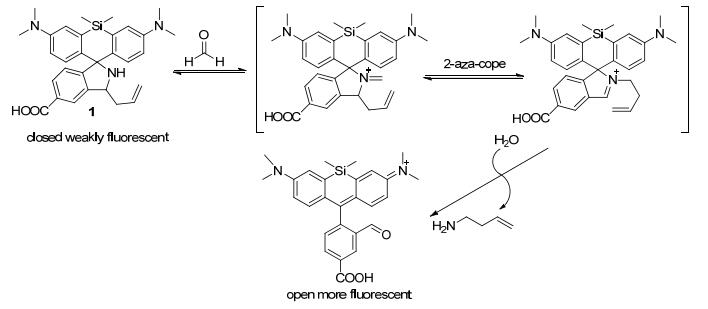
2015年, Chang课题组[20]设计出了一种基于甲醛诱导Aza-cope重排反应的“Turn-on”型荧光探针1, 用于检测细胞内的甲醛.该探针的识别机理如Scheme 2所示.探针1以氨甲基硅罗丹明染料为母体, 分子内有一个关闭的五元环, 表现出较弱的荧光, 与甲醛发生反应后, 探针1发生Aza-cope重排, 最终发生水解, 得到一种具有较强荧光的目标产物, 能够达到对甲醛分子的可视化识别.在磷酸盐缓冲溶液(pH=7.4) 缓冲溶液中, 探针1表现出良好的水溶性以及微弱的荧光, 荧光量子产率(Ф)为0.36, 加入甲醛之后, 在645 nm处可以看到荧光强度增强, 这是因为甲醛的加入导致五元环的开裂, 从而生成荧光较强的目标产物.探针1对甲醛分子的检测具有高选择性和高灵敏性, 此外, 探针1不仅可以检测外源性甲醛, 同时可以检测人体肾上腺细胞(HEK293T)等细胞中赖氨酸特异性蛋白酶去甲基化产生的内源性甲醛.因此, 探针1在生物体内检测甲醛分子具有潜在的应用价值, 为生物体在遗传, 衰老和疾病等生理过程中探测活性羰基化合物提供了一种研究手段.
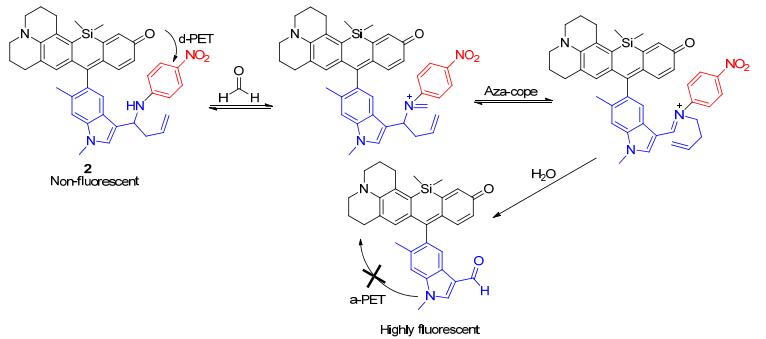
同样地, Chan课题组[21]基于硅罗丹明为骨架, 也设计合成了一个“Turn-on”型荧光探针2, 用于检测细胞内的甲醛(如Scheme 3所示).探针2由三部分组成:黑色部分为荧光团, 蓝色部分为甲醛分子的识别基团, 基于Aza-cope重排反应, 红色部分则代表着荧光猝灭基团.荧光猝灭基团的引入, 使分子中存在光诱导电子转移过程(d-PET), 使得探针2本身没有显示出荧光信号, 与甲醛发生Aza-cope重排反应之后, 水解得到的目标产物中猝灭基团离去, a-PET过程被中断, 表现出较强的荧光.探针2具有良好的光物理性质, 对甲醛分子的检测表现出高选择性和高灵敏性, 探针2可用于人胚肾细胞(HEK293TN)和脑神经瘤细胞(Neuroscreen-1) 活细胞成像.除此之外, 探针2显示出优异的光稳定性, 作者进行了时间推移成像实验并且可以实时捕获探针2和甲醛之间的反应.因此, 探针2为甲醛在活细胞正常以及病理过程方面的研究提供了便利.
2016年, Zeng和Sheng等[22]报道了基于萘为母体的荧光探针3(如Eq. 1所示).探针3在DMF-PBS缓冲溶液(V:V=1:4, pH=7.4) 中, 加入甲醛分子之后, 溶液颜色发生改变, 从无色变为黄色, 能达到肉眼识别, 并且在513 nm处的荧光强度升高, 这主要是由于加入甲醛分子之后, 发生Aza-cope重排反应, 生成的目标产物分子内出现push-pull (推拉)电子效应, 导致分子内电荷转移, 从而引起明显的荧光变化.探针3在pH=3~10.5范围内对甲醛分子的选择性较好, 最小的检测限浓度为0.57 μmol/L.探针3能用于食物里面甲醛的定量检测, 也能用于Hela细胞中甲醛分子的活细胞成像.
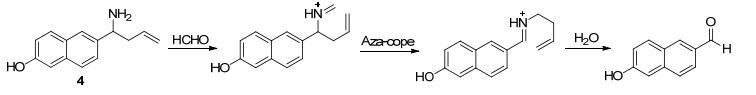
Lin课题组[23]报道了一种基于6-羟基萘为荧光团的甲醛比率型荧光探针4(如Scheme 4所示), 该探针在磷酸盐缓冲溶液(25 mmol/L, pH=7.4, 含1%的丙酮)中, 加入甲醛之后, 探针4在359 nm处的荧光强度呈现降低的趋势, 451 nm处的荧光强度显示增加, I451/I359增加53.2倍, 实现对甲醛的识别.该探针在pH=7.4的磷酸盐缓冲溶液中的检测限为5.96×10-5 mol/L, 在pH=4.5的PBS缓冲溶液中低至1.87×10-5 mol/L.作者用理论计算验证了探针4与甲醛分子的作用机理, 并且把探针4用于HeLa细胞的成像, 结果表明探针4能用于水溶液以及生物样品中甲醛分子的检测.
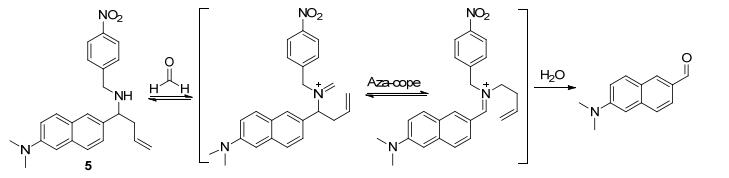
在活细胞成像方面, 双光子荧光探针的优点十分显著, 双光子荧光成像主要通过双光子显微镜来完成.科学实验证实, 设计合成一个好的双光子荧光探针可以从以下几个方面着手, 即在近红外区域拥有大的双光子吸收截面、在水中具有较好的溶解性、对光稳定以及能快速特异性识别某些受体分子[24]. Yuan和Zhang等[25]设计合成了一种双光子荧光探针5(如Scheme 5所示), 以萘衍生物为荧光团, 硝基苯为荧光猝灭基团, 胺为甲醛的识别基团.该探针在PBS-DMSO (V:V=19:1, pH=7.4) 缓冲溶液中表现出较弱的荧光, 加入甲醛分子, 3 h之后, 526 nm处的荧光增强95倍, 这主要是由于与甲醛反应生成的目标产物, 具有D-π-A结构, 从而表现出一种turn-on型荧光反应.该探针在pH=2~10范围内对甲醛分子具有较高的选择性, 检测限低至0.20 μmol/L.除此之外, 探针5具有较低的细胞毒性以及良好的光稳定性, 可以用于内源性以及外源性生物体内甲醛的检测和研究, 因此, 探针5在生物体内检测甲醛分子具有广泛的应用价值.
同样地, Sun和Zhu课题组[26]也报道了另外一种新型双光子荧光探针6(如Eq. 2所示), 该探针以具有较大的斯托克位移以及较好的光稳定性的双光子荧光发射的4-羟基1, 8-萘二甲酰亚胺[27]为母体, 通过Duff反应在羟基的邻位引入醛基, 进而引入甲醛的识别基团.探针6在PBS缓冲溶液(10 mmol/L, pH=7.4, 含0.5%的二甲亚砜)中, 在448 nm处有较强的紫外-可见吸收峰, 荧光发射波长为550 nm, Ф为0.469, 加入甲醛之后, 最大吸收峰变为415 nm, 最大发射波长为520 nm, 均发生蓝移, Ф为0.74.荧光强度与甲醛浓度(0~0.5 mmol/L)之间存在很好的线性关系, 检测限低至5 μmol/L.经实验证明, 探针6可用于HeLa细胞中甲醛的成像, 也可用于活体斑马鱼(zebrafish)中甲醛分子的成像研究.
光动力治疗对于肿瘤的治疗具有准确性高、治疗范围可以控制以及对人体产生的毒副作用较小等优势, 迅速成为当前生物医学领域的研究方向.但是, 我们知道光的穿透力有限, 所以除了胃肠道这些可以利用内窥镜进入的部位的癌症或皮肤癌等可以使用光疗技术, 在其他癌症的诊断和治疗方面, 光动力疗法的使用受到明显的限制[28].目前, 医学成像领域开发了一种常用的新技术, 即正电子发射断层扫描(PET)技术, 这种技术主要通过放射性核素产生正电子, 比较常见的就是放射性标记的18F-脱氧葡萄糖(FDG), 它是产生切伦科夫辐射的理想光源.葡萄糖所具有的结构与FDG相差不大, 于是, FDG在追踪能量代谢异常高的癌细胞中具有一定的应用, 在PET技术中FDG已经普遍用于肿瘤成像[29]. Wilson和Chang课题组[30]设计了一个用于检测甲醛的探针7 (Eq. 3), 该探针与甲醛发生Aza-cope重排反应产生放射性标记的18F-脱氧葡萄糖(FDG), 从而可以实现在活细胞以及生物体内检测甲醛分子, 这一工作为未来在光动力治疗领域的研究和发展荧光探针提供了依据和线索.
1.2 基于探针分子中NHNH2取代基与甲醛发生反应
1, 8-萘二甲酰亚胺衍生物分子中存在较大的共轭体系, 具有良好的光化学和热稳定性、高的荧光量子产率、大的斯托克位移、化学可修饰性强、相对长的激发波长和发射波长以及双光子荧光等[31]优点, 常被用于设计荧光探针的母体. 2016年, Lin课题组[32]以1, 8-萘二甲酰亚胺为母体, NHNH2作为甲醛的反应位点, 设计出了一种基于光诱导电子转移(PET)机理的荧光探针8(如Eq. 4所示).相对于Aza-cope重排反应, NHNH2作为甲醛的反应位点具有快速检测甲醛并且克服了在生理条件下反应达到平衡等缺点.探针8在PBS缓冲溶液(10 mmol/L, pH=7.4, 含1%的二甲亚砜)中, 加入甲醛, 在543 nm处的荧光强度增加325倍, 检测限为7.1×10-7 mol/L, 探针8与甲醛分子的反应速率为0.39 min-1.相对于其他生物体内的活性羰基化合物, 探针8对甲醛具有较高的选择性.探针8在HeLa细胞成像中几乎观察不到荧光, 加入甲醛后, 可以看到明亮的细胞荧光成像.除此之外, 还可以用于生物组织中内源性甲醛的成像研究.
同年, 采用相同的识别机理, 通过改变N-上面的取代基, Lin和Kin等[33]合成了以1, 8-萘酰亚胺为荧光团, 生物素(Biotin)作为取代基的化合物9和氢原子为取代基的化合物10(如Eq. 5所示), 分别用于检测甲醛分子.探针9连有生物素, 相对于探针10而言, 9的生物相容性好, 作者主要研究了探针9的性质.在磷酸盐缓冲溶液中, 加入甲醛后, 探针9在541 nm处的荧光增强140倍.在pH为4~7的变化范围内, 探针9对甲醛分子具有较高的灵敏度, 检测限为0.78 mmol/L.探针9在中性或弱酸性环境的使用范围, 为其在癌细胞中检测甲醛提供了可能.除此之外, 探针9可以选择性的靶向定位于生物素阳性受体(4T-1细胞), 这项研究为探针9在肿瘤部位跟踪内源性甲醛奠定了基础.
溶酶体作为一种细胞器, 主要参与细胞凋亡、物质代谢、细胞膜循环以及酶的形成等生命过程[34].设计合成能够定位某个特定细胞器的荧光探针近年来备受关注[35].吗啉基团的pKa值大约为5~6, 溶酶体的pH范围为4~5, 吗啉质子化之后常存在于溶酶体中, 因此, 吗啉用于溶酶体的定位基团被广泛应用[36].同样地, 2016年, Lin等[37]也报道了以1, 8-萘二甲酰亚胺为母体, NHNH2作为甲醛的反应位点, 并连接常规的吗啉基团作为溶酶体的定位基团的荧光探针11.探针11与甲醛的反应机理如Eq. 6所示, NH2取代基与甲醛发生反应后, 形成C=N双键, 给电子能力降低, PET过程被抑制, 从而荧光恢复.加入甲醛后, 在PBS缓冲溶液(10 mmol/L, pH=7.4, 含1%的二甲亚砜)中, 543 nm处的荧光增强大约350倍, 检测限为5.02×10-6 mol/L.探针11对于甲醛的检测具有较高的选择性, 对于其他的物质, 如乙二醛、甲基乙二醛、对羟基苯甲醛等活性羰基物质均不会产生干扰.探针11对甲醛的反应速率为0.37 min-1, 适用于生物系统的实时监测.探针11主要集中在溶酶体中, 并实现了HeLa细胞中甲醛分子的检测.
采用相同的识别机理, Li和Yu课题组[38]设计了由8-羟基喹啉和苯并噻唑通过共轭组成的荧光探针12(如图12所示).探针12在水溶液中发出很微弱的荧光, 加入甲醛后, 在467 nm处的荧光增强5.5倍, 检测限低至900 nmol/L.作者通过理论计算进一步验证了探针12对甲醛分子的响应变化机制.
1.3 基于探针分子中NH2取代基与甲醛发生的反应
Yoon等[39]报道了以BODIY为荧光团的荧光探针13(如Eq. 8所示), 探针13与甲醛反应之前, 由于分子内存在PET过程, 使探针分子中的荧光团的荧光发生猝灭, 加入甲醛之后, NH2取代基与甲醛发生反应, 形成C=N双键, 给电子能力降低, 使PET过程被中断, 从而目标产物在pH为8的甲酸溶液中, 于535 nm处的荧光增强, 探针13对甲醛的检测限为165 nmol/L.这是一种首次报道的基于BODIY为荧光团在中性环境中对于甲醛分子的检测属于“Turn-on”型响应的荧光探针.
2016年, Lin课题组[40]采用相同的识别机理, 基于1, 8-萘酰亚胺为母体合成了用来识别甲醛的探针14(如Eq. 9所示), 作者通过改变母体分子中4位上的取代基, 探究取代基的变化对探针分子的光谱性质的影响, 当1, 8-萘酰亚胺4位上被NH取代时, 分子内存在光诱导电子转移(PET), 从而使得探针14在乙腈(含5%的HOAc)溶液中, 表现出较弱的荧光, Ф为0.0073, 加入甲醛, 4 min之后, 512 nm处的荧光强度显著增加, 与其他识别甲醛的探针相比较而言, 探针14具有响应快速等特点.探针14的合成与设计为后期甲醛类探针分子的进一步发展打下了很好的基础.
2016年, 与探针14的结构以及识别机理类似, Sun课题组[41]采用1, 8-萘酰亚胺为荧光团, 连接NH2作为甲醛的识别基团, 合成了探针15(如Eq. 10所示).作者探讨了1, 8-萘酰亚胺4位上的取代基, 发现化合物15相对于其他化合物来说, 对甲醛具有较好的响应.探针15对甲醛的检测能实现肉眼识别, 并表现出较好的选择性, 检测限低至0.104 μmol/L.
Thammakhet等[42]基于5-氨基荧光素为荧光基团, 连接聚乙烯醇长链, 利用NH2与甲醛发生反应, 设计合成了一种探针16(如Eq. 11所示).相对于其它用于检测甲醛的探针而言, 探针16具有合成简单, 原料便宜易得, 并且连接聚乙烯醇长链使其具有一定的水溶性, 操作简便等优点.探针16荧光强度在甲醛浓度范围为5.0~100.0 μg·L-1时, 表现出良好的线性关系, 检测限为(3.820±0.079) μg·L-1, 除此之外, 探针16对甲醛的检测表现出较好的重现性(RSD<2.5%).探针16能用于蔬菜、水果以及海鲜中甲醛含量的检测, 还不能用于活细胞中甲醛的检测.
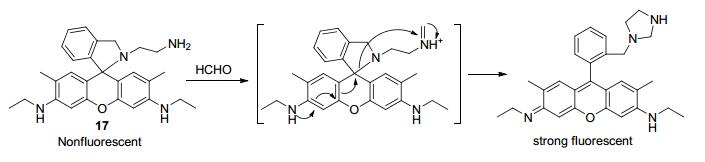
Lin课题组[43]以罗丹明6G为荧光团, 采用两步反应合成了化合物17, 如Scheme 6所示.探针17分子内存在一个封闭的五元环, 表现出较弱的荧光, 与甲醛发生反应后, 五元环被破坏, 目标产物的荧光恢复, 从而实现对甲醛分子的识别.在磷酸盐缓冲溶液(25 mmol/L, pH=7.4, 含50%的二甲亚砜)中, 探针17的荧光量子产率为0.21, 加入一倍甲醛分子后, 在560 nm处的荧光增强7.4倍, 溶液颜色发生变化(无色变为粉色), 探针17对甲醛分子具有较好的选择性和灵敏度, 检测限为7.7×10-7 mol/L.像大多数探针一样, 探针17可以用于HeLa活细胞中甲醛的成像研究, 也可用于像干香菇等食物中甲醛的识别, 并且是首个能实现对甲醛气体的裸眼识别的荧光探针, 为生物系统、食品工业以及生活环境中甲醛分子的检测提供了指导.
1.4 金属配合物用于检测甲醛的荧光探针
2014年, Li等[44]基于金属镝(Dy)和2-氨基-1, 4-苯二甲酸(H2aBDC)合成了大小可控的有机金属配位聚合物(Dy2aBDC3). Dy2aBDC3聚合物粒子无论是在固态还是分散在DMF, 乙醇, 丙酮等溶剂中都没有荧光, 但是, 当有少量的水存在时, 聚合物显示出较强的荧光; 当聚合物分散在含有醛类化合物像甲醛、乙醛和丙醛的DMF溶液中时, 在365 nm波长的紫外灯下照射, 可观测到荧光强度增强, 随着醛类化合物的含量的增高, 荧光强度不断增强并且发生不同程度的红移.从诱导发生红移的程度相比较而言, Dy2aBDC3聚合物粒子对于甲醛的灵敏度要高于乙醛和丙醛.
Fang课题组[45]通过“点击化学”设计合成了化合物18 (Eq. 12所示), 分子中杯[4]芳烃为母体, 1, 2, 3-三唑为连接基团和识别基团, 双-硝基苯并噁二唑(NBD)为荧光发射基团.在化合物18的THF溶液中对Ag+的识别具有较高的选择性, 不断改变加入Ag+的浓度, 可以发现, 化合物在527 nm处的发射峰不断降低, 并且在576 nm处出现一个新的吸收峰.化合物18对Ag+的检测限为6.2×10-7 mol/L.考虑到甲醛是一种强还原剂, 能快速的把Ag配合物中的配位中心Ag+还原为Ag0.作者向18-Ag+混合溶液中不断改变加入甲醛的浓度, 可以发现, 在576 nm处的荧光吸收峰不断降低, 在527 nm处出现新的吸收峰. 18-Ag+混合溶液对HCHO的识别的检测限低至1 μmol/L.这一配位和还原过程可以被重复至少五次左右.
1.5 其他机理检测甲醛
2015年, Wong等[46]主要通过Au(Ⅲ)化合物催化三组分偶合反应(A3-coupling reaction)设计了一种新型的快速检测HCHO的方法.这种检测方法的机理为连接树脂的胺、甲醛以及连有荧光团的端炔在Au(Ⅲ)催化作用下发生三组分偶合反应, 生成具有荧光的目标化合物, 从而达到对HCHO的识别, 机理如Eq. 13所示.
Liu课题组[47]利用HCHO能将Ag+还原成Ag, 设计了20~30 nm的Au核-银壳纳米离子(Au@AgNRs), Au@AgNRs通过荧光光谱法和比色法选择性地检测甲醛分子.在AuNRs-Ag+-甘氨酸-氢氧化钠缓冲溶液中, AuNRs在748和555 nm处出现吸收带, 利用HCHO将Ag+还原为Ag0包裹在AuNRs的表面上以形成Au@Ag沉淀, 随着甲醛的加入, 748 nm处的吸收带不断发生红移, 并且溶液颜色也在不断发生变化. Au核-Ag壳纳米离子(Au@AgNRs)对甲醛的检测具有较高的选择性, 检测限低至6.3×10-11 g/mL. Au@AgNRs纳米粒子广泛应用于水样中甲醛的检测, 并且展现出广泛的实用性.
2 检测甲基乙二醛的荧光探针
甲基乙二醛作为生物体内活性羰基化合物的一种, 伴随着脂质过氧化、非酶糖基化等能量代谢过程而产生[48].甲基乙二醛有较高的反应活性, 易与生物大分子如DNA, 蛋白质等物质的氨基和巯基发生反应, 生成糖基化终产物(ACEs)[49].研究表明, 人体内的糖尿病、心血管疾病[50]、痛觉过敏[51]以及肾脏疾病[52]均与甲基乙二醛含量的升高有较大的关系.近年来, 科研工作者对甲基乙二醛的研究已经有了许多进展, 但甲基乙二醛在细胞过程中的作用和发病机制仍然很少被了解[4a].因此, 发展和研究用于生物体系中甲基乙二醛含量的检测和成像成为当前研究的热点.目前, 关于甲基乙二醛荧光探针的报道主要是基于邻苯二甲胺识别基团与甲基乙二醛发生的反应.
2013年, Spiegel等[53]首次报道了3种基于BODIY为母体, 连接邻苯二甲胺作为识别基团, 通过改变BODIY和邻苯二甲胺上面的取代基用于检测甲基乙二醛的荧光探针19~21, 如Eq. 14所示.探针19~21的识别机理主要是由于探针分子内的邻苯二甲胺基团与BODIY基团之间存在a-PET过程, 当邻苯二甲胺基团与甲基乙二醛反应后, 这一过程被抑制, 从而使目标化合物的荧光恢复.作者通过理论计算预测到探针19与甲基乙二醛反应后发生强烈的荧光增强, 探针20其次, 探针21产生较小的变化.实验表明, 探针19与甲基乙二醛反应后, 荧光强度增加10倍, Ф由0.003增加为0.326, 而探针20仅发生中等强度的荧光增强, 对于探针21而言, 响应很小.于是, 作者主要对探针19做了进一步研究.在PBS缓冲溶液中, 探针19与甲基乙二醛共存1 h的检测限为50~100 nmol/L, 并且共存的时间越长, 检测限也越高.探针19与甲基乙二醛的一阶速率常数为4.56×10-3 s-1.探针19不仅适用于HeLa活细胞中甲基乙二醛分子的成像研究, 同时, 也能基于高效液相色谱法应用于血清或血浆中甲基乙二醛浓度的检测, 为后期各种疾病的诊断提供了便利.
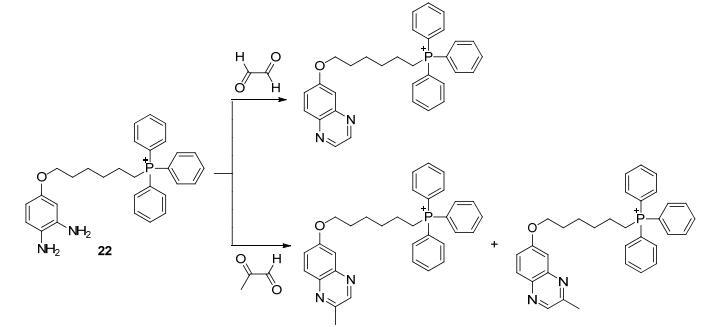
三苯基膦正离子带正电荷, 线粒体膜外带负电荷, 因此三苯基膦取代基作为线粒体的定位基团, 常被用于设计合成某些生物小分子的荧光探针[54]. Murphy等[55]采用邻苯二甲胺作为甲基乙二醛和乙二醛的反应位点, 连接三苯基膦定位基团合成了探针22(如Scheme 7所示).作者发现, 化合物22对甲基乙二醛的反应活性和灵敏度要高于乙二醛, 这与甲基乙二醛对生物体的毒性要强于乙二醛的说法一致.除此之外, 作者通过探针22研究了细胞体内甲基乙二醛与乙二醛的变化与线粒体指出了方向.
基于上述相同的识别机理, 2015年, Zhou课题组[56]采用4-溴-1, 8-萘二甲酸酐为原料, 首次设计合成了一个用于检测甲基乙二醛的双光子荧光探针23(如Eq. 15所示).在PBS缓冲溶液(10 mmol/L, pH=7.4, 含10%的二甲亚砜)中, 探针23几乎没有荧光, 加入甲基乙二醛后, 在528 nm处荧光增强33倍, Ф由0.029变为0.256.在甲基乙二醛浓度范围为0~10 μmol/L范围内, 探针23的荧光强度与甲基乙二醛的浓度呈现出良好的线性关系.探针23对甲基乙二醛的检测具有较高的选择性和灵敏性, 检测限为77 nmol/L.除此之外, 探针23可以用于HeLa活细胞中甲基乙二醛分子检测的双光子成像研究.
超分子水凝胶具有生物相容性和可降解性等[57]优势, 被广泛用于伤口愈合[58], 药物释放[59]以及组织工程等[60]领域, 近年来, Liang及其课题组[61]设计合成了基于联吡啶的超分子凝胶用于可以检测Cd2+, 并且伴随着选择性好和灵敏度高的特征, 为这一技术在分析检测领域的应用提供了基础.类似地, 2016年, Liang等[62]基于邻苯二甲胺与甲基乙二醛具有较高的反应活性, 设计合成了一种水凝胶前体24 (如Eq. 16所示), 可以原位点击(in situ “click”)与甲基乙二醛发生反应形成化合物25, 然后化合物25自组装形成超分子水凝胶, 从而达到原位检测和可视化识别, 并且作者设计合成的化合物24对甲基乙二醛的识别不受NO的干扰, 具有较高的选择性.
Kool课题组[63]基于荧光腙设计合成了探针26(如Eq. 17所示), 用于甲基乙二醛的标记和检测.荧光腙分子的设计需满足以下几个条件:在环境条件下, 对水具有较高的稳定性, 不易发生水解; 荧光发生完全猝灭, 几乎观察不到背景的荧光; 必须是热力学不稳定的腙, 从而能驱使反应的发生.探针26分子设计中2-肼基吡啶作为亲核试剂, 实现对甲基乙二醛分子的识别, 偶氮苯作为荧光猝灭基团, 5-甲氧基邻氨基苯甲酸作为催化剂.在磷酸盐缓冲溶液中, 探针26与甲基乙二醛反应后, 荧光强度增强30倍.作者通过催化剂的选择, 底物范围以及醛的结构等方面对这一反应进程的影响进行了探讨.探针26适用于活细胞中甲基乙二醛分子的成像研究.
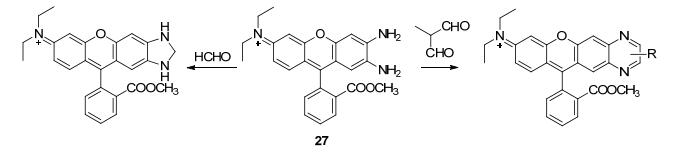
2017年, Zeng等[64]基于邻二氨基罗丹明母体设计合成了探针27(如Scheme 8所示), 可以实现对甲醛“Turn-ON”型以及对甲基乙二醛“Turn-OFF”型的检测.在Tris-HCl缓冲溶液(10 mmol/L, pH=7.4, 含30%的乙醇)中, 探针27在642 nm, 处几乎没有荧光, 加入甲醛后, 发射波长蓝移至620 nm, Ф由0.07增加为0.55, 探针27对甲醛分子的检测限为8.3 μmol/L.加入甲基乙二醛之后, 探针27的发射波长红移至642 nm, 荧光强度降低, Ф由0.07降低为0.0014.同样地, 探针27也能用于活细胞中实现对甲醛“Turn-ON”型以及对甲基乙二醛“Turn-OFF”型的成像研究.
钌和铱的配合物因具有较好的光物理性质和电化学性质, 常被用来作为生物小分子以及离子的探针[65]. 2017年, Zhang课题组[66]基于邻苯二甲胺取代的菲咯啉和联吡啶报道了一种钌的配合物28和一种铱的配合物29, 用于对甲基乙二醛分子的检测, 如Eq. 18所示.由于探针分子内存在光诱导电子转移(PET)过程, 使其在磷酸盐缓冲溶液中表现出较弱的荧光, 探针28和29的荧光量子产率分别为0.12%和0.23%;与甲基乙二醛反应后, PET过程被抑制, 探针在pH 3~11范围内, 表现出强烈的荧光.化合物28和29对甲基乙二醛的检测均具有一定的选择性, 探针28的检测限为0.78 μmol/L, 化合物29为1.15 μmol/L.作者通过密度泛函理论对这一识别机理进行了计算.除此之外, 探针28和29具有较好的生物相容性, 可以成功地用于RAW 264.7巨噬细胞和D. magna中的成像研究.
3 检测丙二醛的荧光探针
氧化应激(Oxidative Stress)作为生命体内的一种非健康状态, 主要是通过生命体内氧化过程及抗氧化系统失衡所引起, 这一状态与多种疾病, 如心血管疾病、肿瘤及糖尿病等的产生和发展有着不可分割的联系[67].丙二醛(Malondialdehyde, MDA)作为生物体内活性羰基化合物的一种类型, 是氧化应激的一种经典的生物标志物[68], 生物体内氧化应激的失衡程度可以通过机体内丙二醛含量的高低体现出来[69], 于是, 丙二醛也可以作为一种重要的病理指标, 当生物体内, 丙二醛含量的升高会引起一序列的疾病, 如白血病、糖尿病、癌症、心血管疾病、哮喘以及动脉粥样硬化肝脏疾病等[70].
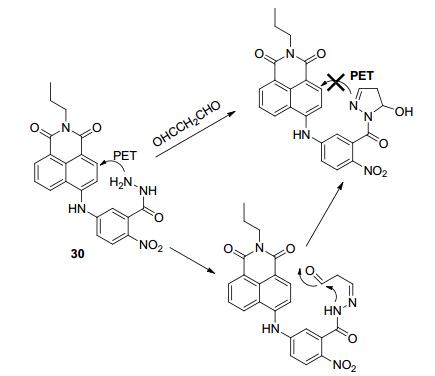
2015年, Liu课题组[71]以可与MDA作用的苯甲酰肼为起始物, 通过对反应机理的深入研究构建了可用于体内成像的荧光探针30 (Scheme 9), 首次实现了MDA在活细胞内的成像. Gleiter课题组[72]报道2, 4-二硝基苯甲酰胺由于取代基硝基具有强的吸电子效应, 使得其与MDA具有较高的反应活性, 于是, 作者通过改变苯甲酰肼上取代基的位置和类别, 探索出了与MDA反应活性较高的苯甲酰肼, 作为MDA的识别基团, 然后通过NH基团将1, 8-萘酰亚胺荧光基团连接起来, 设计合成了探针30.探针30分子内存在PET过程, 在DMSO-磷酸盐缓冲溶液(V:V=1:9, 50 mmol/L, pH=7.4) 中, 探针30表现出较弱的荧光, 与MDA发生反应后, PET过程被抑制, 在553和520 nm处荧光分别增强174倍和52倍.探针30对MDA的识别具有较高的选择性, 检测限低至0.6 μmol/L.探针30首次实现了MDA在活细胞内的成像研究, 它不仅可以检测内源性的MDA, 还可以实时追踪肿瘤细胞的内源性MDA及氧化应激状态下MDA的发生.这一工作促进了后期氧化应激相关病理过程的探究及抗氧化药物的开发.
4 检测丙烯醛的荧光探针
丙烯醛是一种高毒性的不饱和醛, 它可以通过油、木炭、木材、塑料和其他有机物质的不完全燃烧而产生, 也可以通过多胺、脂质、氨基酸和其他生物分子的氧化应激下内源产生[73].丙烯醛具有高反应性, 常会引发许多免疫疾病和炎症等[74], 被认为是对人类健康的真正危害.发展新型的、有效的用来检测丙烯醛的方法显得至关重要.
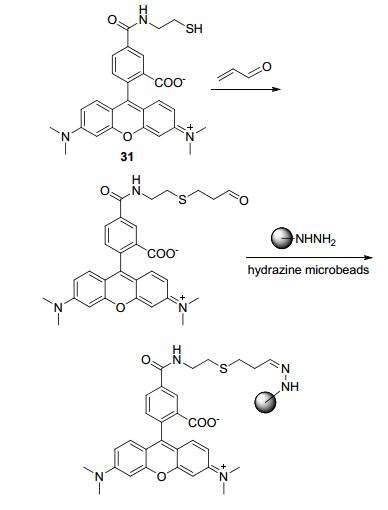
2014年, Nagano等[75]基于四甲基羧基罗丹明为母体设计合成了荧光探针31(如Scheme 10所示), 用于快速检测血浆里面的丙烯醛.探针与丙烯醛的识别过程分两步进行, 首先探针31与含丙烯醛的血浆发生迈克尔加成反应, 然后加成产物与附有聚乙二醇且含有NHNH2官能团的复合微球作用, 从而通过共价键将探针分子连接在复合微球上.复合微球与含有丙烯醛的溶液混合后, 表现出较强的荧光, 并且其他活性羰基化合物产生的干扰小.这种方法与传统的检测方法相比, 具有操作简单、便宜、实用、灵敏度高等优势, 检测限为0.54 μmol/L.作者指出这一方法潜在的缺陷是探针31对丙烯醛的检测并不能从严格意义上与其他某些活性羰基化合物区分开, 但是这不能阻碍它在疾病诊断以及临床上面的应用.
5 总结与展望
活性羰基化合物作为生物体内重要的活性物种之一, 其在人体内的含量发生异常时, 会引发许多疾病, 设计合成用于快速、高效、实时监测羰基化合物含量的探针在生理, 病理以及疾病诊断和控制方面的研究具有重要的意义.本文根据活性羰基化合物的种类, 归纳和总结了近年来用于检测甲醛、甲基乙二醛、丙二醛以及丙烯醛等荧光探针的设计, 识别机理以及应用.通过综述发现, 目前关于活性羰基化合物的研究主要集中在甲醛和甲基乙二醛, 这是因为目前发现了较多对甲醛或甲基乙二醛物种的特异性有机反应, 而其它活性羰基化合物物种的特异性有机反应相对较少.所以, 今后应当寻找某种新的用来特异性识别某种活性羰基化合物的有机反应.除此之外, 部分探针存在不能用于微生物环境, 选择性不好, 灵敏性不高, 发射未达到近红外区等不足, 今后设计新的用来识别活性羰基化合物的荧光探针可以从以下几个方面进行优化:选择近红外发射的荧光团; 与目标化合物结合之后显示“Switch-ON”型荧光响应或者比率型荧光探针; 探针与活性羰基化合物选择性识别之后, 产物的荧光强度要高, 荧光量子产率也应该较好; 探针具有较低的细胞毒性和较好的生物相容性, 最好是水溶性或两亲性的荧光探针; 探针与某种活性羰基化合物的识别具有较高的灵敏性和选择性; 进一步开发和设计出双光子, 近红外还能定位在某一细胞器中的选择性荧光探针, 从而实现细胞器的功能成像.还有一点, 目前荧光探针的成像研究主要进行到细胞成像阶段, 少部分进行到活体组织成像阶段.今后设计出能对生物体进行原位、实时、动态的可视化监测的高选择性和灵敏度的活性羰基化合物荧光探针, 开拓其在疾病诊断和临床方面的应用已成为主要研究方向, 也是当今化学与生命科学研究的重点和难点.
-
-
[1]
(a) Kalapos, M. P. Diabetes Res. Clin. Pract. 2013, 99, 260.
(b) Onyango, A. N. Chem. Phys. Lipids 2012, 165, 777. -
[2]
Semchyshyn, H. M. Sci. World J. 2014, 2014, 10. http://www.ncbi.nlm.nih.gov/pubmed/24634611
-
[3]
(a) Colzani, M.; De Maddis, D.; Casali, G.; Carini, M.; Vistoli, G.; Aldini, G. ChemMedChem 2016, 11, 1778.
(b) Regazzoni, L.; De Courten, B.; Garzon, D.; Altomare, A.; Marinello, C.; Jakubova, M.; Vallova, S.; Krumpolec, P.; Carini, M.; Ukropec, J.; Ukropcova, B.; Aldini, G. Sci. Rep. 2016, 6, 27224.
(c) Hwang, S.; Lee, Y.-M.; Aldini, G.; Yeum, K.-J. Molecules 2016, 21, 280.
(d) Biswas, S.; Mano, J. I. Plant Cell Physiol. 2016, 57, 1432. -
[4]
(a) Nemet, I.; Varga-Defterdarović, L.; Turk, Z. Mol. Nutr. Food Res. 2006, 50, 1105.
(b) Kato, S.; Burke, P. J.; Koch, T. H.; Bierbaum, V. M. Anal. Chem. 2001, 73, 2992.
(c) Soman, A.; Qiu, Y.; Chan Li, Q. J. Chromatogr. Sci. 2008, 46, 461. -
[5]
(a) Jung, H. S.; Chen, X.; Kim, J. S.; Yoon, J. Chem. Soc. Rev. 2013, 42, 6019.
(b) Niu, L.-Y.; Chen, Y.-Z.; Zheng, H.-R.; Wu, L.-Z.; Tung, C.-H.; Yang, Q.-Z. Chem. Soc. Rev. 2015, 44, 6143.
(c) Wang, S.; Shen, S.; Zhang, Y.; Dai, X.; Zhao, B. Chin. J. Org. Chem. 2014, 34, 1717(in Chinese).
(王胜清, 申世立, 张延如, 戴溪, 赵宝祥, 有机化学, 2014, 34, 1717.) -
[6]
(a) Santos-Figueroa, L. E.; Moragues, M. E.; Climent, E.; Agostini, A.; Martinez-Manez, R.; Sancenon, F. Chem. Soc. Rev. 2013, 42, 3489.
(b) Li, B.; Meng, W.; Yang, M.; Su, N.; Liu, X.; Yang, B. Chin. J. Org. Chem. 2015, 35, 2629(in Chinese).
(李波, 孟文斐, 杨美盼, 苏娜, 刘晓静, 杨秉勤, 有机化学, 2015, 35, 2629.) -
[7]
(a) Guo, Z.; Park, S.; Yoon, J.; Shin, I. Chem. Soc. Rev. 2014, 43, 16.
(b) Wang, X.-D.; Wolfbeis, O. S. Chem. Soc. Rev. 2014, 43, 3666.
(c) Zhang, R.; Zhao, J.; Han, G.; Liu, Z.; Liu, C.; Zhang, C.; Liu, B.; Jiang, C.; Liu, R.; Zhao, T.; Han, M.-Y.; Zhang, Z. J. Am. Chem. Soc. 2016, 138, 3769. -
[8]
Chen, X.; Tian, X.; Shin, I.; Yoon, J. Chem. Soc. Rev. 2011, 40, 4783. doi: 10.1039/c1cs15037e
-
[9]
(a) Zhang, H.; Liu, R.; Liu, J.; Li, L.; Wang, P.; Yao, S. Q.; Xu, Z.; Sun, H. Chem. Sci. 2016, 7, 256.
(b) Meimetis, L. G.; Giedt, R. J.; Mikula, H.; Carlson, J. C.; Kohler, R. H.; Pirovich, D. B.; Weissleder, R. Chem. Commun. 2016, 52, 9953. -
[10]
(a) Chen, X.; Lee, K.-A.; Ren, X.; Ryu, J.-C.; Kim, G.; Ryu, J.-H.; Lee, W.-J.; Yoon, J. Nat Protoc. 2016, 11, 1219.
(b) Fan, J.; Han, Z.; Kang, Y.; Peng, X. Sci. Rep. 2016, 6, 19562.
(c) Zhu, X.; Xiong, M.; Liu, H.-W.; Mao, G.-J.; Zhou, L.; Zhang, J.; Hu, X.; Zhang, X.-B.; Tan, W. Chem. Commun. 2016, 52, 733. -
[11]
(a) Choi, S.-A.; Park, C. S.; Kwon, O. S.; Giong, H.-K.; Lee, J.-S.; Ha, T. H.; Lee, C.-S. Sci. Rep. 2016, 6, 26203.
(b) Li, W.; Fang, B.; Jin, M.; Tian, Y. Anal. Chem. 2017, 89, 2553.
(c) Xu, K.; Luan, D.; Wang, X.; Hu, B.; Liu, X.; Kong, F.; Tang, B. Angew. Chem. 2016, 128, 12943. -
[12]
(a) Deng, B.; Ren, M.; Wang, J.-Y.; Zhou, K.; Li, W. Sens. Actuators, B 2017, 248, 50.
(b) Cheng, D.; Pan, Y.; Wang, L.; Zeng, Z.; Yuan, L.; Zhang, X.; Chang, Y.-T. J. Am. Chem. Soc. 2017, 139, 285.
(c) Li, Y.; Lü, Z.; Liu, M.; Xing. G. Chin. J. Org. Chem. 2016, 36, 962(in Chinese).
(李杨洁, 吕子奇, 刘敏, 邢国文, 有机化学, 2016, 36, 962.) -
[13]
(a) Yuan, Q.; Zhao, Z.-M.; Zhang, Y.-R.; Su, L.; Miao, J.-Y.; Zhao, B.-X. Sens. Actuators, B. 2017, 247, 736.
(b) Wang, Y.; Li, J.; Feng, L.; Yu, J.; Zhang, Y.; Ye, D.; Chen, H.-Y. Anal. Chem. 2016, 88, 12403.
(c) Li, M.; Wang, Y.; Liu, G.; Lü, H.; Xing, G. Chin. J. Org. Chem. 2017, 37, 356(in Chinese).
(李美含, 王宇童, 刘广建, 吕海娟, 邢国文, 有机化学, 2017, 37, 356.) -
[14]
(a) Zhang, J.-X.; Li, H.; Chen, C.-F.; Lan, R.; Chan, W.-L.; Law, G.-L.; Wang, W.-K.; Wang, K.-L. Chem. Commun. 2012, 48, 9646.
(b) Wang, B.; Fan, J.; Wang, X.; Zhu, H.; Wang, J.; Mu, H.; Peng, X. Chem. Commun. 2015, 51, 792.
(c) Singh, H.; Lee, H. W.; Heo, C. H.; Byun, J. W.; SarKar, A. R.; Kim, H. M. Chem. Commun. 2015, 51, 12099. -
[15]
(a) Shi, Y.; Lan, F.; Matson, C.; Mulligan, P.; Whetstine, J. R.; Cole, P. A.; Casero, R. A.; Shi, Y. Cell 2004, 119, 941.
(b) Tsukada, Y.-I.; Fang, J.; Erdjument-Bromage, H.; Warren, M. E.; Borchers, C. H.; Tempst, P. and Zhang, Y. Nature 2006, 439, 811.
(c) Whetstine, J. R.; Nottke, A.; Lan, F.; Huarte, M.; Smolikov, S.; Chen, Z.; Spooner, E.; Li, E.; Zhang, G.; Colaiacovo, M. Cell 2006, 125, 467. -
[16]
Tong, Z.; Han, C.; Luo, W.; Wang, X.; Li, H.; Luo, H.; Zhou, J.; Qi, J.; He, R. Age 2013, 35, 583. doi: 10.1007/s11357-012-9388-8
-
[17]
Tong, Z.; Zhang, J.; Luo, W.; Wang, W.; Li, F.; Li, H.; Luo, H.; Lu, J.; Zhou, J.; Wan, Y.; He, R. Neurobiol. Aging 2011, 32, 31. doi: 10.1016/j.neurobiolaging.2009.07.013
-
[18]
Tulpule, K.; Dringen, R. J. Neurochem. 2013, 127, 7. doi: 10.1111/jnc.12356/full
-
[19]
(a) Goodman, C. G.; Johnson, J. S. J. Am. Chem. Soc. 2015, 137, 14574.
(b) Overman, L. E.; Humphreys, P. G.; Welmaker, G. S. Org. React. 2011, 748.
(c) Winter, R. F.; Rauhut, G. Chem.-Eur. J. 2002, 8, 641.
(d) Mccormack, M. P.; Shalumova, T.; Tanski, J. M.; Waters, S. P. Org. Lett. 2010, 12, 3906. -
[20]
Brewer, T. F.; Chang, C. J. J. Am. Chem. Soc. 2015, 137, 10886. doi: 10.1021/jacs.5b05340
-
[21]
Roth, A.; Li, H.; Anorma, C.; Chan, J. J. Am. Chem. Soc. 2015, 137, 10890. doi: 10.1021/jacs.5b05339
-
[22]
Xu, J.; Zhang, Y.; Zeng, L.; Liu, J.; Kinsella, J. M.; Sheng, R. Talanta 2016, 160, 645. doi: 10.1016/j.talanta.2016.08.010
-
[23]
He, L.; Yang, X.; Liu, Y.; Kong, X.; Lin, W. Chem. Commun. 2016, 52, 4029. doi: 10.1039/C5CC09796G
-
[24]
(a) Dai, Z.-R.; Ge, G.-B.; Feng, L.; Ning, J.; Hu, L.-H.; Jin, Q.; Wang, D.-D.; Lv, X.; Dou, T.-Y.; Cui, J.-N. J. Am. Chem. Soc. 2015, 137, 14488.
(b) Zhu, B.; Li, P.; Shu, W.; Wang, X.; Liu, C.; Wang, Y.; Wang, Z.; Wang, Y.; Tang, B. Anal. Chem. 2016, 88, 12532.
(c) Zhou, L.; Zhang, X.; Wang, Q.; Lv, Y.; Mao, G.; Luo, A.; Wu, Y.; Wu, Y.; Zhang, J.; Tan, W. J. Am. Chem. Soc. 2014, 136, 9838.
(d) Zhang, J.; Zhu, X.-Y.; Hu, X.-X.; Liu, H.-W.; Li, J.; Feng, L. L.; Yin, X.; Zhang, X.-B.; Tan, W. Anal. Chem. 2016, 88, 11892.
(e) Bae, S. K.; Heo, C. H.; Choi, D. J.; Sen, D.; Joe, E.-H.; Cho, B. R.; Kim, H. M. J. Am. Chem. Soc. 2013, 135, 9915.
(f) Cheng, D.; Pan, Y.; Wang, L.; Zeng, Z.-B.; Yuan, L.; Zhang, X.-B.; Chang, Y.-T. J. Am. Chem. Soc. 2017, 139, 285.
(g) Kim, H. M.; Cho, B. R. Acc. Chem. Res. 2009, 42, 863. -
[25]
Li, J.-B.; Wang, Q.-Q.; Yuan, L.; Wu, Y.-X.; Hu, X.-X.; Zhang, X.-B.; Tan, W. Analyst 2016, 141, 3395. doi: 10.1039/C6AN00473C
-
[26]
Xie, Z.; Ge, J.; Zhang, H.; Bai, T.; He, S.; Ling, J.; Sun, H.; Zhu, Q. Sens. Actuators, B 2017, 241, 1050. doi: 10.1016/j.snb.2016.10.039
-
[27]
Qian, X.; Xiao, Y.; Xu, Y.; Guo, X.; Qian, J.; Zhu, W. Chem. Commun. 2010, 46, 6418 doi: 10.1039/c0cc00686f
-
[28]
Ethirajan, M.; Chen, Y.; Joshi, P.; Pandey, R. K. Chem. Soc. Rev. 2011, 40, 340. doi: 10.1039/B915149B
-
[29]
Kotagiri, N.; Sudlow, G. P.; Akers, W. J.; Achilefu, S. Nat. Nanotechnol. 2015, 10, 370. doi: 10.1038/nnano.2015.17
-
[30]
Liu, W.; Truillet, C.; Flavell, R. R.; Brewer, T. F.; Evans, M. J.; Wilson, D. M.; Chang, C. J. Chem. Sci. 2016, 7, 5503. doi: 10.1039/C6SC01503D
-
[31]
(a) Han, Q.; Mou, Z.; Wang, H.; Tang, X.; Dong, Z.; Wang, L.; Dong, X.; Liu, W. Anal. Chem. 2016, 88, 7206.
(b) Shu, W.; Yan, L.; Liu, J.; Wang, Z.; Zhang, S.; Tang, C.; Liu, C.; Zhu, B.; Du, B. Ind. Eng. Chem. Res. 2015, 54, 8056.
(c) Zhou, P.; Yao, J.; Hu, G.; Fang, J. ACS Chem. Biol. 2016, 11, 1098.
(d) Liu, X.-L.; Du, X.-J.; Dai, C.-G.; Song, Q.-H. J. Org. Chem. 2014, 79, 9481. -
[32]
Tang, Y.; Kong, X.; Liu, Z.-R.; Xu, A.; Lin, W. Anal. Chem. 2016, 88, 9359. doi: 10.1021/acs.analchem.6b02879
-
[33]
Lee, Y. H.; Tang, Y.; Verwilst, P.; Lin, W.; Kim, J. S. Chem. Commun. 2016, 52, 11247. doi: 10.1039/C6CC06158C
-
[34]
(a) Watts, C. Biochim. Biophys. Acta, Proteins Proteomics 2012, 1824, 14.
(b) Zhao, H. Traffic 2012, 13, 1307. -
[35]
Zhu, H.; Fan, J.; Du, J.; Peng, X. Acc. Chem. Res. 2016, 49, 2115. doi: 10.1021/acs.accounts.6b00292
-
[36]
(a) Zhou, J.; Shi, W.; Li, L.; Gong, Q.; Wu, X.; Li, X.; Ma, H. Anal. Chem. 2016, 88, 4557.
(b) Dong, B.; Song, X.; Wang, C.; Kong, X.; Tang, Y.; Lin, W. Anal. Chem. 2016, 88, 4085.
(c) Qiu, K.; Huang, H.; Liu, B.; Liu, Y.; Huang, Z.; Chen, Y.; Ji, L.-N.; Chao, H. ACS Appl. Mater. Interfaces. 2016, 8, 12702.
(d) Wan, Q.; Chen, S.; Shi, W.; Li, L.; Ma, H. Angew. Chem., Int. Ed. 2014, 53, 10916.
(e) Wang, Y.; Li, J.; Feng, L.; Yu, J.; Zhang, Y.; Ye, D.; Chen, H.-Y. Anal. Chem. 2016, 88, 12403.
(f) Fan, J.; Han, Z.; Kang, Y.; Peng, X. Sci. Rep. 2016, 6, 1. -
[37]
Tang, Y.; Kong, X.; Xu, A.; Dong, B.; Lin, W. Angew. Chem., Int. Ed. 2016, 55, 3356. doi: 10.1002/anie.201510373
-
[38]
Liu, C.; Shi, C.; Li, H.; Du, W.; Li, Z.; Wei, L.; Yu, M. Sens. Actuators, B 2015, 219, 185. doi: 10.1016/j.snb.2015.04.131
-
[39]
Song, H.; Rajendiran, S.; Kim, N.; Jeong, S. K.; Koo, E.; Park, G.; Thangadurai, T. D.; Yoon, S. Tetrahedron Lett. 2012, 53, 4913. doi: 10.1016/j.tetlet.2012.06.117
-
[40]
Dong, B.; Song, X.; Tang, Y.; Lin, W. Sens. Actuators, B 2016, 222, 325. doi: 10.1016/j.snb.2015.07.039
-
[41]
Liu, C.; Cheng, A.-W.; Xia, X.-K.; Liu, Y.-F.; He, S.-W.; Guo, X.; Sun, J.-Y. Anal. Methods 2016, 8, 2764. doi: 10.1039/C6AY00108D
-
[42]
Aksornneam, L.; Kanatharana, P.; Thavarungkul, P.; Thammakhet, C. Anal. Methods 2016, 8, 1249. doi: 10.1039/C5AY02719E
-
[43]
He, L.; Yang, X.; Ren, M.; Kong, X.; Liu, Y.; Lin, W. Chem. Commun. 2016, 52, 9582. doi: 10.1039/C6CC04254F
-
[44]
Zhao, X. J.; Yang, J. H.; Li, Y. F. Sens. Actuators, B 2014, 203, 417. doi: 10.1016/j.snb.2014.06.095
-
[45]
Zhang, S.; Yang, H.; Ma, Y.; Fang, Y. Sens. Actuators, B 2016, 227, 271. doi: 10.1016/j.snb.2015.12.016
-
[46]
Wong, K.-F.; Deng, J.-R.; Wei, X.-Q.; Shao, S.-P.; Xiang, D.-P.; Wong, M.-K. Org. Biomol. Chem. 2015, 13, 7408. doi: 10.1039/C5OB00966A
-
[47]
Lin, J.-M.; Huang, Y.-Q.; Liu, Z.-B.; Lin, C.-Q.; Ma, X.; Liu, J.-M. RSC Adv. 2015, 5, 99944. doi: 10.1039/C5RA16266A
-
[48]
Thornalley, P. J. Drug Metabol. Drug Interact. 2008, 23, 125. http://europepmc.org/abstract/med/18533367
-
[49]
(a) Matafome, P.; Sena, C.; Seiça, R. Endocrine 2013, 43, 472.
(b) Degen, J.; Vogel, M.; Richter, D.; Hellwig, M.; Henle, T. J. Agric. Food Chem. 2013, 61, 10253. -
[50]
Kilhovd, B. K.; Juutilainen, A.; Lehto, S.; Rönnemaa, T.; Torjesen, P. A.; Hanssen, K. F.; Laakso, M. Atherosclerosis 2009, 205, 590. doi: 10.1016/j.atherosclerosis.2008.12.041
-
[51]
Eberhardt, M. J.; Filipovic, M. R.; Leffler, A.; De La Roche, J.; Kistner, K.; Fischer, M. J.; Fleming, T.; Zimmermann, K.; IvanovicBurmazovic, I.; Nawroth, P. P. J. Biol. Chem. 2012, 287, 28291. doi: 10.1074/jbc.M111.328674
-
[52]
Nakayama, K.; Nakayama, M.; Iwabuchi, M.; Terawaki, H.; Sato, T.; Kohno, M.; Ito, S. Am. J. Nephrol. 2008, 28, 871. doi: 10.1159/000139653
-
[53]
Wang, T.; Douglass Jr, E. F.; Fitzgerald, K. J.; Spiegel, D. A. J. Am. Chem. Soc. 2013, 135, 12429. doi: 10.1021/ja406077j
-
[54]
(a) Hou, J.-T.; Wu, M.-Y.; Li, K.; Yang, J.; Yu, K.-K.; Xie, Y.-M.; Yu, X.-Q. Chem. Commun. 2014, 50, 8640.
(b) Yuan, H.; Cho, H.; Chen, H. H.; Panagia, M.; Sosnovik, D. E.; Josephson, L. Chem. Commun. 2013, 49, 10361.
(c) Xiao, H.; Li, J.; Zhao, J.; Yin, G.; Quan, Y.; Wang, J.; Wang, R. J. Mater. Chem. B 2015, 3, 1633.
(d) Li, P.; Zhang, W.; Li, K.; Liu, X.; Xiao, H.; Zhang, W.; Tang, B. Anal. Chem. 2013, 85, 9877.
(e) Masanta, G.; Heo, C. H.; Lim, C. S.; Bae, S. K.; Cho, B. R.; Kim, H. M. Chem. Commun. 2012, 48, 3518.
(f) Lim, C. S.; Masanta, G.; Kim, H. J.; Han, J. H.; Kim, H. M.; Cho, B. R. J. Am. Chem. Soc. 2011, 133, 11132. -
[55]
Pun, P. B. L.; Logan, A.; Darley-Usmar, V.; Chacko, B.; Johnson, M. S.; Huang, G. W.; Rogatti, S.; Prime, T. A.; Methner, C.; Krieg, T.; Fearnley, I. M.; Larsen, L.; Larsen, D. S.; Menger, K. E.; Collins, Y.; James, A. M.; Kumar, G. D. K.; Hartley, R. C.; Smith, R. a. J.; Murphy, M. P. Free Radicals Biol. Med. 2014, 67, 437. doi: 10.1016/j.freeradbiomed.2013.11.025
-
[56]
Tang, T.; Zhou, Y.; Chen, Y.; Li, M.; Feng, Y.; Wang, C.; Wang, S.; Zhou, X. Anal. Methods 2015, 7, 2386. doi: 10.1039/C4AY02922D
-
[57]
Mei, B.; Miao, Q.; Tang, A.; Liang, G. Nanoscale 2015, 7, 15605. doi: 10.1039/C5NR04563K
-
[58]
Yang, Z.; Liang, G.; Ma, M.; Abbah, A. S.; Lu, W. W.; Xu, B. Chem. Commun. 2007, 8, 843. http://www.ncbi.nlm.nih.gov/pubmed/17308650
-
[59]
Su, T.; Tang, Z.; He, H.; Li, W.; Wang, X.; Liao, C.; Sun, Y.; Wang, Q. Chem. Sci. 2014, 5, 4204. doi: 10.1039/C4SC01603C
-
[60]
Yu, Y.; Chau, Y. Biomacromolecules 2015, 16, 56. doi: 10.1021/bm501063n
-
[61]
Miao, Q.; Wu, Z.; Hai, Z.; Tao, C.; Yuan, Q.; Gong, Y.; Guan, Y.; Jiang, J.; Liang, G. Nanoscale 2015, 7, 2797. doi: 10.1039/C4NR06467D
-
[62]
Liu, S.; Luo, Y.; Liang, G. Nanoscale 2016, 8, 766. doi: 10.1039/C5NR07179H
-
[63]
Yuen, L. H.; Saxena, N. S.; Park, H. S.; Weinberg, K.; Kool, E. T. ACS Chem. Biol. 2016, 11, 2312. doi: 10.1021/acschembio.6b00269
-
[64]
Liu, C.; Jiao, X.; He, S.; Zhao, L.; Zeng, X. Dyes Pigm. 2017, 138, 23. doi: 10.1016/j.dyepig.2016.11.020
-
[65]
(a) Zhang, F.; Liang, X.; Zhang, W.; Wang, Y.-L.; Wang, H.; Mohammed, Y. H.; Song, B.; Zhang, R.; Yuan, J. Biosens. Bioelectron. 2017, 87, 1005.
(b) Cao, L.; Zhang, R.; Zhang, W.; Du, Z.; Liu, C.; Ye, Z.; Song, B.; Yuan, J. Biomaterials 2015, 68, 21.
(c) Zhang, R.; Ye, Z.; Song, B.; Dai, Z.; An, X.; Yuan, J. Inorg. Chem. 2013, 52, 10325.
(d) Zhang, R.; Ye, Z.; Yin, Y.; Wang, G.; Jin, D.; Yuan, J.; Piper, J. A. Bioconjugate Chem. 2012, 23, 725. -
[66]
Zhang, W.; Zhang, F.; Wang, Y.-L.; Song, B.; Zhang, R.; Yuan, J. Inorg. Chem. 2017, 56, 1309. doi: 10.1021/acs.inorgchem.6b02443
-
[67]
Gorrini, C.; Harris, I. S. and Mak, T. W. Nat. Rev. Drug Discovery 2013, 12, 931. doi: 10.1038/nrd4002
-
[68]
Riggins, J. N.; Pratt, D. A.; Voehler, M.; Daniels, J. S.; Marnett, L. J. J. Am. Chem. Soc. 2004, 126, 10571. doi: 10.1021/ja040010q
-
[69]
Niedernhofer, L. J.; Daniels, J. S.; Rouzer, C. A.; Greene, R. E.; Marnett, L. J. J. Biol. Chem. 2003, 278, 31426. doi: 10.1074/jbc.M212549200
-
[70]
(a) Jain, S. K.; Mcvie, R.; Smith, T. Diabetes Care 2000, 23, 1389.
(b) Tajika, K.; Okamatsu, K.; Takano, M.; Inami, S.; Yamamoto, M.; Murakami, D.; Kobayashi, N.; Ohba, T.; Hata, N.; Seino, Y.; Mizuno, K. Circ. J. 2012, 76, 2211.
(c) Romieu, I.; Barraza-Villarreal, A.; Escamilla-Nuñez, C.; Almstrand, A.-C.; Diaz-Sanchez, D.; Sly, P. D.; Olin, A.-C. J. Allergy Clin. Immunol. 2008, 121, 903.
(d) Dillioglugil, M. O.; Mekık, H.; Muezzinoglu, B.; Ozkan, T. A.; Demir, C. G.; Dillioglugil, O. Int. Urol. Nephrol. 2012, 44, 1691. -
[71]
Chen, J.; Zeng, L.; Xia, T.; Li, S.; Yan, T.; Wu, S.; Qiu, G.; Liu, Z. Anal. Chem. 2015, 87, 8052. doi: 10.1021/acs.analchem.5b02032
-
[72]
Pilz, J.; Meineke, I.; Gleiter, C. H. J. Chromatogr. Biomed. Appl. 2000, 742, 315. doi: 10.1016/S0378-4347(00)00174-2
-
[73]
Hensley, K.; Robinson, K. A.; Gabbita, S. P.; Salsman, S.; Floyd, R. A. Free Radic. Biol. Med. 2000, 28, 1456. doi: 10.1016/S0891-5849(00)00252-5
-
[74]
(a) Sharmin, S.; Sakata, K.; Kashiwagi, K.; Ueda, S.; Iwasaki, S.; Shirahata, A.; Igarashi, K. Biochem. Biophys. Res. Commun. 2001, 282, 228.
(b) Yoshida, M.; Tomitori, H.; Machi, Y.; Hagihara, M.; Higashi, K.; Goda, H.; Ohya, T.; Niitsu, M.; Kashiwagi, K.; Igarashi, K. Biochem. Biophys. Res. Commun. 2009, 378, 313. -
[75]
Togashi, M.; Terai, T.; Kojima, H.; Hanaoka, K.; Igarashi, K.; Hirata, Y.; Urano, Y.; Nagano, T. Chem. Commun. 2014, 50, 14946. doi: 10.1039/C4CC02578D
-
[1]
-
-

 扫一扫看文章
扫一扫看文章
计量
- PDF下载量: 62
- 文章访问数: 3779
- HTML全文浏览量: 1103


 下载:
下载:









 下载:
下载:

