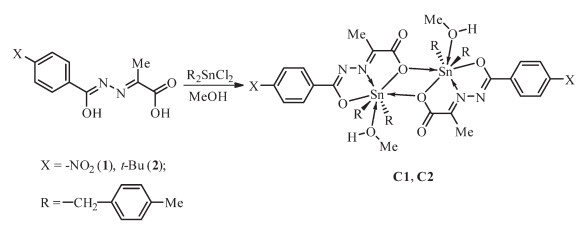
 图1
配合物的合成
Figure1.
Synthesis of the complexes
图1
配合物的合成
Figure1.
Synthesis of the complexes

Citation: ZHANG Zhi-Jian, JIANG Wu-Jiu, LIU Yang, KUANG Dai-Zhi, YU Jiang-Xi, ZHU Xiao-Ming, TAN Yu-Xing. Syntheses, Crystal Structures and Biological Activity of N-(2-Propionic acid)-aroyl Hydrazone Di-p-methylbenzytin Complexes[J]. Chinese Journal of Inorganic Chemistry, 2017, 33(9): 1603-1610. doi: 10.11862/CJIC.2017.198

N-(2-丙酸)-芳甲酰腙二对甲基苄基锡配合物的合成、晶体结构及生物活性
English
Syntheses, Crystal Structures and Biological Activity of N-(2-Propionic acid)-aroyl Hydrazone Di-p-methylbenzytin Complexes
-
Key words:
- organotin complex
- / hydrazone
- / synthesis
- / crystal structure
- / biological activity
-
0 引言
在20世纪60年代末,顺铂(Ⅱ)抗癌作用的发现及临床应用,开辟了金属配合物抗癌药物研究的新领域。随着人们对金属配合物药理作用认识的进一步深入,新的高效、低毒、具有抗癌活性的金属化合物不断被合成出来[1-4]。自1980年Crowe[5]首次报道二烃基锡化合物具有抑制癌细胞增殖作用以来,有机锡化合物在抗癌药物领域受到了人们的广泛关注[6-8],与其他结构类型的有机锡化合物相比,二烃基锡化合物通常具有更好的抗癌活性[9-10]。因此,利用具有生物活性基团的配体同二烃基锡反应制备新型有机锡配合物并研究其抗癌活性,这一工作就显得更加重要。
酰腙类化合物是由酰肼和醛酮缩合而得的产物,分子中有酰基氧、亚胺基氮和其他取代基上的配位原子,且由于酰腙基团存在酮式和烯醇式,使酰腙化合物表现出多样的配位形式和较强的生物活性[11-13]。近年来,国内外许多研究人员发现酰腙类化合物具有抗癌、杀菌、杀虫、消炎等多种活性[14-16],且其代谢产物均低毒或无毒。
因此,本文采用二对甲基苄基二氯化锡与N-(2-丙酸)-芳甲酰腙反应,合成了2个取代苄基锡配合物,初步研究了配合物对癌细胞及正常人体细胞的体外抑制活性,以及与小牛胸腺DNA的相互作用,为筛选具有更多、更高生物活性以及新型、高效、低毒、广谱的金属抗癌药物提供一定的理论基础。
1 实验部分
1.1 仪器和试剂
IR用日本岛津Prestige-21红外光谱仪(4 000~400 cm-1,KBr压片)测定;1H、13C和119Sn NMR用Bruker AVANCE-500核磁共振仪测定;元素分析用PE-2400(Ⅱ)元素分析仪测定;晶体结构用Bruker SMART APEX Ⅱ CCD单晶衍射仪测定;荧光光谱用日本日立F-7000荧光光谱仪测定;紫外光谱用日本岛津公司UV-2550型紫外-可见光谱仪测定;热重用德国NETZSCH TG 209 F3热重分析仪;熔点用北京泰克X-4双目体视显微熔点测定仪测定(温度计未经校正)。
N-(2-丙酸)-芳甲酰腙和二对甲基苄基二氯化锡参考文献[17-18]方法合成。溴化乙锭(EB)、小牛胸腺DNA、三羟甲基氨基甲烷(Tris)为Sigma-Aldrich公司产品,其它试剂均为分析纯,溶剂参考文献[19]方法纯化,水为超纯水。Tris-HCl(0.01mol·L-1)缓冲溶液通过称取一定量Tris用0.1 mol·L-1的盐酸溶液调至pH值为7.40,使用前配制;小牛胸腺DNA的纯度通过比较260和280 nm处的吸光度来确定(A260/A280=1.8 ~1.9),用所需pH值条件下缓冲溶液配制,浓度通过测定260 nm处的吸光度计算而得(ε260=6 600 L·mol-1·cm-1),其储备液置于4 ℃保存;溴化乙锭溶液通过称取适量溴化乙锭固体,用pH=7.40的Tris-HCl(0.01 mol·L-1)缓冲溶液配制。
1.2 配合物的合成
于50 mL圆底烧瓶中,加入1 mmol N-(2-丙酸)-对硝基苯甲酰腙或N-(2-丙酸)-对叔丁基苯甲酰腙,1 mmol二对甲基苄基二氯化锡,30 mL甲醇,搅拌回流10 h。冷却,过滤,蒸除溶剂,用甲醇重结晶,得淡黄色晶体C1或C2。
配合物C1:产率75%。m.p. 118~120 ℃(dec)。元素分析(C54H58N6O12Sn2):实测值(计算值,%):C,53.21(53.14);H,4.73(4.79);N,6.92(6.89)。IR(KBr,cm-1):3 084,3 022 ν(Ar-H),2 920 ν(C-H),1 637 ν(C=N),1 616,1 327 ν(COO),1 527 ν(C=N-N=C),1 209 ν(C-O),590 ν(Sn-O),555 ν(Sn-O-Sn),503 ν(Sn-N), 460ν(Sn -C)。1H NMR(500 MHz,CDCl3):δ 8.28(d,J=8.7 Hz,2H),8.10(d,J=8.7 Hz,2H),6.83~6.77(m,8H), 3.28 (s,4H),2.15(s,6H),2.07(s,3H)。13C NMR(125 MHz,CDCl3):δ 172.65, 169.20, 153.66, 149.85, 139.14, 135.15, 133.15,129.63,128.91,128.21,123.21,50.82,36.43,20.86,12.94。119Sn NMR(Me4Sn,187 MHz,CDCl3):δ -636.40。
配合物C2:产率71 %。m.p. 118~120 ℃(dec)。元素分析(C31H38N2O4Sn):实测值(计算值,%):C,59.89(59.92);H, 6.17(6.16);N, 4.51(4.51)。IR(KBr, cm-1):3 045,3 020 ν(Ar-H),2 960,2 866 ν(C-H),1 666 ν(C =N),1 606,1 392 ν(COO),1 581 ν(C=N-N=C),1 205 ν(C-O),596 ν(Sn-O),540 ν(Sn-O-Sn),503 ν(Sn-N),460 ν(Sn-C)。1H NMR(500 MHz,CDCl3):δ 7.95(d,J=8.3 Hz,2H),7.47(d,J=8.3 Hz,2H),7.29(d,J=7.8 Hz,1H),7.19(d,J=7.8 Hz,1H),6.87(m,6H),3.10(s,4H),2.20(s,6H),2.11(s,3H),1.38(s,9H)。13C NMR(125 MHz,CDCl3):δ 174.71,155.83,138.48, 135.03, 132.65, 129.27, 129.23,129.10,128.40,127.96,125.20,50.89,35.09,31.20,21.24,20.91,13.06。119Sn NMR(Me4Sn,187 MHz,CDCl3):δ -509.34。
1.3 晶体结构测定
选取尺寸为0.20 mm×0.18 mm×0.18 mm(C1)和0.23 mm×0.21 mm×0.21 mm(C2)的配合物晶体,在Bruker SMART APEX Ⅱ CCD单晶衍射仪上,采用经石墨单色化的Mo Kα射线(λ=0.071 073 nm),以φ~ω扫描方式收集衍射数据。全部数据经Lp因子和多重扫描吸收校正。晶体结构由直接法解出,全部非氢原子坐标在差值Fourier合成中陆续确定,氢原子H6A(C1),H4(C2)为差值Fourier合成给出氢原子在晶胞中的位置坐标,其余为理论加氢法给出氢原子在晶胞中的位置坐标。对氢原子和非氢原子分别采用各向同性和各向异性热参数进行全矩阵最小二乘法修正,全部结构分析计算工作采用SHELX-97程序系统完成[20]。
CCDC:1547502,C1;1547503,C2。
Complex C1 C2 Empirical formula C54H58N6O12Sn2 C31H38N2Sn Formula weight 1 220.44 621.32 T/K 296(2) 296(2) Crystal system Triclinic Monoclinic Space group P1 P21/n a/nm 1.074 53(7) 1.829 97(7) b/nm 1.130 91(8) 0.870 01(3) c/nm 1.301 50(9) 1.929 44(7) α/(°) 93.612 0(10) 90 β/(°) 100.471 0(10) 90.596 0(10) γ/(°) 116.373 0(10) 90 Volume/nm3 1.374 70(16) 3.071 68(19) Z 1 4 Dc/(Mg·m-3) 1.474 1.344 Absorption coefficient/mm-1 0.974 0.867 F(000) 620 1280 θ range/(°) 2.61~25.10 3.05~25.10 Limiting indices -12 ≤ h ≤ 12, -13 ≤ k ≤ 13, -15 ≤ l ≤ 15 -21 ≤ h ≤ 21, -10 ≤ k ≤ 10, -23 ≤ l ≤ 23 Reflection collected, unique 12 667, 4 870 (Rint=0.015 4) 38 638, 5 463 (Rint=0.044 7) Completeness 0.994 0.999 Max. and min. transmission 0.844 2 and 0.829 1 0.838 9 and 0.825 5 Data, restraint, parameter 4 870, 0, 342 5 463, 31, 354 Goodness-of-fit on F2 1.027 1.087 Final R indices [I>2σ(I)] R1=0.020 7, wR2=0.053 5 R1=0.040 8, wR2=0.110 8 R indices (all data) R1=0.023 0, wR2=0.054 6 R1=0.047 6, wR2=0.114 1 (Δρ)max, (Δρ)min/(e.nm-3) 397, -259 1 136, -456 1.4 紫外光谱测定
在室温下,用DMSO分别配置浓度为1×10-4 mol·L-1的C1、C2溶液,25 ℃下分别测试它们的紫外-可见吸收光谱,样品测试均使用测日本岛津公司UV-2550型紫外-可见光谱仪。
1.5 体外抗癌活性测定
将待测药物溶于少量DMSO,用水稀释至所需浓度,保持最终wDMSO < 0.1%。MCF7、HepG2、H460细胞株取自美国组织培养库(ATCC),HL-7702细胞株购自中国科学院生物化学与细胞生物学研究所。HL-7702细胞株用含10%胎牛血清,2% L-谷氨酰胺的RPMI 1640(GIBICO公司)培养基,MCF7、HepG2、H460细胞株用含10%胎牛血清的RPMI 1640(GIBICO公司)培养基,在5%(体积分数)CO2、37 ℃饱和湿度培养箱内进行体外培养。体外抗癌药敏试验是通过MTT法测定。数据处理使用Graph Pad Prism version 7.0程序,化合物IC50通过程序中具有S形剂量响应的非线性回归模型进行拟合得到。
1.6 与DNA相互作用实验
在5 mL容量瓶中分别加入小牛胸腺DNA、EB及不同浓度的配合物溶液,混匀,25 ℃下放置3.5 h,分别扫描荧光光谱,激发波长为258 nm,发射波长见图谱,激发和发射光谱扫描狭缝宽度均为5.0 nm。
2 结果与讨论
2.1 谱学研究
从配合物C1和C2的红外谱图中可以看出,C1和C2中羧基的反对称伸缩振动峰和对称伸缩振动峰分别出在1 616,1 385 cm-1和1 606,1 392 cm-1处,反对称伸缩振动频率和对称伸缩振动频率之差分别为231和214 cm-1,表明2个配合物中的羧酸根均是以单齿形式与Sn配位[21]。此外,C1和C2配位键的特征峰ν(Sn-O)、ν(Sn-O-Sn)、ν(Sn-N)和ν(Sn-C)分别位于590、555、503、460 cm-1和596、540、503、460 cm-1处[22-23],表明有机锡配合物的形成, 并且初步证实2个配合物有着相似的化学结构。
在1H NMR谱中,配合物各组峰的积分面积之比与预期结构的各组质子数相对吻合[24];从谱图中可以看到,配合物C1和C2中与锡原子相连的对甲基苄基的亚甲基质子分别出峰在δ=3.28,3.10,其峰形是由一个正常的单峰和一对小卫星峰组成。究其原因,这是由于119Sn-H耦合的结果[25],其耦合常数JSn-H分别为111.0和89.7 Hz;2个配合物的其它氢质子出峰也基本保持一致,进一步说明了2个配合物具有相似的化学结构。在13C NMR谱中,其各组峰与理论推测结构碳原子数相吻合[24],与X射线单晶衍射结果一致。在119Sn NMR谱中,C1和C2分别在-636.40和-509.34处呈现一个单峰,表明2个配合物中均仅存在一种单一的有机锡化合物。
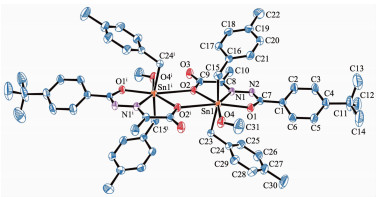
2.2 晶体结构
配合物的主要键长和键角数据列于表 2,分子结构见图 2、3。2个配合物均为双锡核分子,存在1个Sn2O2平面中心四元环,环的中心就是分子的对称中心。四元环由羧基氧原子以μ-桥联配位Sn原子,且与2个锡原子的键长不等, 其中C1中Sn1-O2 0.234 20(14) nm,C2中Sn1-O2 0.232 2(2) nm,均属于正常Sn-O共价键长; 而C1中Sn1-O2i 0.270 3(15) nm,C2中Sn1-O2i 0.283 51(25) nm,大于Sn-O共价键长,但是小于锡原子与氧原子范氏半径之和,比文献报道[26-27]相似配合物的Sn-O略长。
C1 Sn1-C19 0.214 2(2) Sn1-C11 0.214 4(2) Sn1-O1 0.216 66(13) Sn1-N1 0.223 82(16) Sn1-O2 0.234 20(14) Sn1-O6 0.242 26(17) C19-Sn1-C11 164.55(8) C19-Sn1-O1 96.42(7) C11-Sn1-O1 92.01(7) C19-Sn1-N1 96.71(8) C11-Sn1-N1 98.29(7) 01-Sn1-N1 70.56(6) C19-Sn1-O2 91.26(7) C11-Sn1-O2 90.52(7) 01-Sn1-O2 140.14(5) N1-Sn1-O2 69.71(5) C19-Sn1-O6 86.38(9) C11-Sn1-O6 82.83(8) 01-Sn1-O6 77.53(6) N1-Sn1-O6 148.08(6) 02-Sn1-O6 142.15(5) C2 Sn1-C23 0.213 9(4) Sn1-O1 0.214 1(2) Sn1-C15 0.214 6(4) Sn1-N1 0.223 0(3) Sn1-O2 0.232 2(2) Sn1-O4 0.239 4(3) C23-Sn1-O1 97.71(12) C23-Sn1-C15 164.08(16) 01-Sn1-C15 93.66(14) C23-Sn1-N1 97.43(16) 01-Sn1-N1 71.04(10) C15-Sn1-N1 96.78(13) C23-Sn1-O2 88.94(14) 01-Sn1-O2 141.09(9) C15-Sn1-O2 89.17(13) N1-Sn1-O2 70.09(9) C23-Sn1-O4 85.26(17) 01-Sn1-O4 78.98(10) C15-Sn1-O4 86.03(14) N1-Sn1-O4 150.00(10) 02-Sn1-O4 139.91(9) 在配合物C1结构中,Sn1与来自配体中的2个氧原子O1和O2,1个亚氨基氮原子N1,1个配位甲醇氧原子O6,来自2个对甲基苄基中的亚甲基碳原子C11和C19以及来自另一个配体分子中的O2i等配位,形成七配位五角双锥构型。O1、O2、O6、N1、O2i占据了赤道平面的5个位置,2个亚甲基碳原子C11和C19则占据了该平面两侧的轴向位置,轴向C11-Sn1-C19键角为164.55(8)°,比180°小了15.45°,且赤道平面的5个原子与中心锡原子的键长不等(dSn1-O1=0.216 66(13) nm;dSn1-O2=0.234 20(14) nm;dSn1-O6=0.242 26(17) nm;dSn1-N1=0.223 82(16) nm;dSn1-O2i=0.270 3(15) nm),其差值为0.007 16~0.053 64 nm,且键角也不相等(∠O1-Sn1-O6=77.53(6)°;∠O1-Sn1-N1=70.56(6)°;∠N1-Sn1-O2=69.71(5)°;∠O2-Sn1-O2i=65.27°;∠O6-Sn1-O2i=76.95°),因此该配合物中心锡原子为七配位畸变五角双锥构型。配合物C2与C1分子结构类似,键参数差异不大,中心锡原子也为七配位畸变五角双锥构型。在2个配合物结构中, Sn-N键长为:C1:0.223 82(16) nm,C2:0.223 0(3) nm,与文献报道相似[28-29]。
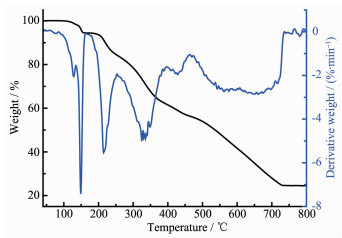
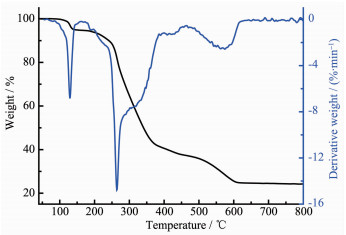
2.3 热稳定性研究
为了研究配合物的热稳定性,采用NETZSCH TG 209 F3热重分析仪,在空气氛下,加热速度为20 ℃·min-1,气体流速为20 mL·min-1,在40~800 ℃范围内对配合物进行热重测试。如图 4、5所示,随温度的升高,配合物发生相似的失重过程。在初始阶段40~170 ℃,配合物C1失重为5.52%(理论值:5.24%),C2为5.26%(理论值:5.15%),分别对应配合物失去2个配位甲醇分子;配合物C1、C2的中间失重阶段均无明显的平台阶段,在170~800 ℃范围内连续失重,对应配合物分子失去2个N-(2-丙酸)-芳甲酰腙配体及4个对甲基苄基,最终稳定在约24.55%(C1)和24.23%(C2),残余物与SnO2的计算含量24.58%(C1)及24.14%(C2)吻合。上述热分析结果表明配合物C1、C2分别在120、135 ℃之前可稳定存在。
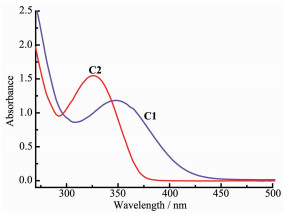
2.4 紫外光谱
配合物C1、C2的紫外-可见吸收光谱如图 6所示。配合物C1、C2分别在348和326 nm处有较大吸收,推测其原因,是由于配合物C1中苯环上的取代基(硝基)是生色基团,使得其吸收峰向长波方向移动;而C2中苯环上取代基(叔丁基)中C-H与苯环产生超共轭效应,也使苯环的吸收带红移,吸收强度增大,但是这种助色作用相对较弱[30]。
2.5 体外抗癌活性研究
表 3列出了配合物C1、C2和卡铂对体外培养癌细胞NCI-H460(人肺癌细胞)、HepG2(人肝癌细胞)、MCF7(人乳腺癌细胞)以及HL7702(正常人体肝细胞)的抑制活性。从表中数据可知,配合物C1、C2对3种癌细胞都有较好的抑制作用,尤其是配合物C2对H460和MCF7的抑制作用均优于卡铂,对HepG2的抑制作用与卡铂相当;在对正常人体细胞毒性试验中,配合物C1、C2对HL7702细胞毒性均大于卡铂,但配合物C2对HL7702的细胞毒性远小于C1,故配合物C2有望进一步化学优化作为抗癌药物的候选化合物;分析配合物C1、C2在正常人体细胞毒性试验中差异原因可能与配体分子中苯环上取代基有关[31-32]。
Complex IC50/(μmol·L-1) H460 HepG2 MCF7 HL7702 C1 8.18±0.39 5.89±0.17 10.23±0.92 11.73±1.09 C2 4.18±0.13 1.89±0.55 2.41±0.51 6.61±0.33 Carboplatin 6.26±1.02 1.70±0.32 8.22±1.29 21.38±2.41 2.6 配合物与DNA-EB作用的荧光光谱研究
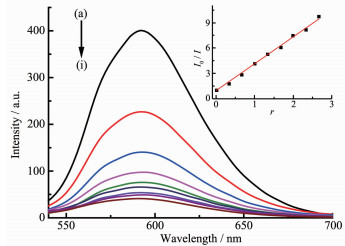
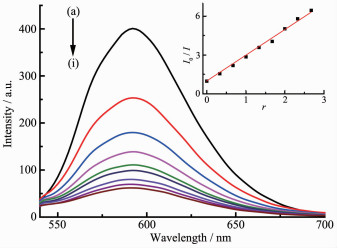
溴化乙锭(EB)是一种荧光染料,但其本身的荧光很弱。在DNA溶液中,EB能平行地嵌入到双螺旋DNA内部的碱基对之间,从而使荧光显著增强。当配合物与EB的DNA溶液共存时,便会发生竞争反应,配合物可能把EB从DNA双螺旋中挤出,导致荧光强度发生猝灭,因而EB可用作DNA结构的荧光探针[33]。
图 7、8分别为不同浓度的配合物C1及C2对EB-DNA复合体系的荧光淬灭曲线。加入配合物C1或C2后,DNA-EB体系的荧光均明显降低,说明配合物C1或C2的存在使DNA-EB体系的荧光产生了猝灭。根据经典Stern-Volmer方程[34]:I0/I=1+Ksqr,由曲线拟合推断其作用属于静态猝灭[35],分别计算出配合物C1、C2与DNA作用的猝灭常数Ksq为3.16和1.99,比文献[36-37]报道的结合常数大,其大小定量地反映出配合物与DNA插入作用的能力。通过比较结合常数可以看出配合物C1、C2与DNA存在较强的插入作用,推测可能是配合物的中心锡原子与DNA分子中的碱基基团配位结合,配合物中的端基配体芳环插入到DNA的碱基对中,竞争了EB与DNA的结合,把EB从DNA分子的碱基对中挤出。结合配合物对癌细胞的体外抑制活性分析,配合物C1、C2的抗肿瘤活性与DNA的结合能力相关,杀死肿瘤细胞很可能是通过配体和有机锡的协同效应[38]与DNA相互键合所致[33]。
3 结论
二对甲基苄基二氯化锡分别与N-(2-丙酸)-对硝基苯甲酰腙及N-(2-丙酸)-对叔丁基苯甲酰腙反应,合成了2个取代苄基锡配合物(C1、C2)。结构分析表明,两个配合物分子均为双锡核分子,以Sn2O2四元环为中心对称,且锡原子与配位原子形成七配位畸变五角双锥构型。热分析结果表明,在空气氛下,配合物C1在120 ℃、C2在135 ℃以下可稳定存在。研究了配合物C1、C2对癌细胞H460、HepG2、MCF7以及正常人体肝细胞HL7702的体外抑制活性,结果表明配合物C1、C2对3种癌细胞都有较好的抑制作用,并且C2对HL7702的细胞毒性远小于C1,故配合物C2有望作为抗癌药物的候选化合物;在Tris-HCl缓冲溶液中,以EB作为荧光探针,用荧光光谱法初步研究了配合物与小牛胸腺DNA的相互作用,结果表明配合物C1、C2与小牛胸腺DNA作用是插入结合作用所致。
Supporting information is available at http://www.wjhxxb.cn
-
-
[1]
Albert J, Bosque R, Crespo M, et al. Dalton Trans., 2015, 44(30):13602-13614 doi: 10.1039/C5DT01713K
-
[2]
Fang S, Chen L, Yu M, et al. Org. Biomol. Chem., 2015, 13(16):4714-4726 doi: 10.1039/C5OB00007F
-
[3]
Zou B Q, Lu X, Qin Q P, et al. RSC Adv., 2017, 7(29):17923-17933 doi: 10.1039/C7RA00826K
-
[4]
Yan B R, Lü X Y, Du H, et al. Chem. Pap.-Chem. Zvesti, 2016, 70(7):983-993
-
[5]
Crowe A J, Smith P J. Atassi Ghanem. Chem.-Biol. Interact., 1980, 32(1/2):171-178
-
[6]
Xuan H, Zhang J, Wang Y, et al. Bioorg. Med. Chem. Lett., 2016, 26(2):570-574 doi: 10.1016/j.bmcl.2015.11.072
-
[7]
Salam M A, Hussein M A, Ramli I, et al. J. Organomet. Chem., 2016, 813:71-77 doi: 10.1016/j.jorganchem.2016.04.007
-
[8]
Tabassum S, Yadav S, Arjmand F. Inorg. Chim. Acta, 2014, 423B:38-45
-
[9]
Sirajuddin M, Ali S, McKee V, et al. RSC Adv., 2014, 4(101):57505-57521 doi: 10.1039/C4RA10487K
-
[10]
Roy M, Roy S, Singh K S, et al. New J. Chem., 2016, 40(2):1471-1484 doi: 10.1039/C5NJ02637G
-
[11]
Ferreira I P, Piló E D L, Recio-Despaigne A A, et al. Bioorg. Med. Chem., 2016, 24(13):2988-2998 doi: 10.1016/j.bmc.2016.05.007
-
[12]
Mohanraj M, Ayyannan G, Raja G, et al. J. Photochem. Photobiol. B, 2016, 158:164-173 doi: 10.1016/j.jphotobiol.2016.03.005
-
[13]
Sedaghat T, Ebrahimi Y, Carlucci L, et al. J. Organomet. Chem., 2015, 794:223-230 doi: 10.1016/j.jorganchem.2015.06.034
-
[14]
韩杰, 周晓霞, 陈思宝, 等.有机化学, 2014, 34(4):741-748 http://www.cnki.com.cn/Article/CJFDTOTAL-SYQY201603027.htmHAN Jie, ZHOU Xiao-Xia, CHEN Si-Bao, et al. Chin. J. Org. Chem., 2014, 34(4):741-748 http://www.cnki.com.cn/Article/CJFDTOTAL-SYQY201603027.htm
-
[15]
任争, 康玉华, 石贞玉, 等.药学学报, 2010, 45(9):1109-1115 http://www.cnki.com.cn/Article/CJFDTOTAL-SYQY201603027.htmREN Zheng, KANG Yu-Hua, SHI Zhen-Yu, et al. Acta Pharm. Sin., 2010, 45(9):1109-1115 http://www.cnki.com.cn/Article/CJFDTOTAL-SYQY201603027.htm
-
[16]
Foscolos A S, Papanastasiou I, Foscolos G B, et al. Med. Chem. Commun., 2016, 7(6):1229-1236 doi: 10.1039/C6MD00035E
-
[17]
何水样, 曹文凯, 陈军利, 等.高等学校化学学报, 2002, 23(6):991-995 http://www.cnki.com.cn/Article/CJFDTOTAL-SYQY201603027.htmHE Shui-Yang, CAO Wen-Kai, CHEN Jun-Li, et al. Chem. J. Chinese Universities, 2002, 23(6):991-995 http://www.cnki.com.cn/Article/CJFDTOTAL-SYQY201603027.htm
-
[18]
Sisido K, Takeda Y, Kinugawa Z, et al. J. Am. Chem. Soc., 1961, 83(3):538-541 doi: 10.1021/ja01464a008
-
[19]
Armarego W L F, Chai C L L. Purification of Laboratory Chemicals. 6th Ed. Oxford:Butterworth-Heinemann, 2009.
-
[20]
Sheldrick G M. SHELXTL Version 5.03, Siemens Analytical X-ray Division, Madison, Wisconsin, USA, 1994.
-
[21]
Deacon G B, Phillips R J. Coord. Chem. Rev., 1980, 33:227-250 doi: 10.1016/S0010-8545(00)80455-5
-
[22]
Jiang W J, Yang N F, Kuang D Z, et al. Chin. J. Struct. Chem., 2011, 30(9):1327-1331
-
[23]
Yang Y, Hong M, Xu L, et al. J. Organomet. Chem., 2016, 804:48-58 doi: 10.1016/j.jorganchem.2015.12.041
-
[24]
Pretsch E, Buhlmann P, Martin B. Structure Determination of Organic Compounds:Tables of Spectral Data. 4th Ed. Berlin and Heidelberg:Springer-Verlag, 2009.
-
[25]
Hong M, Yin H, Chen S, et al. J. Organomet. Chem., 2010, 695(5):653-662 doi: 10.1016/j.jorganchem.2009.11.035
-
[26]
Baba H S, Dietz C, Lutter M, et al. Organometallics, 2015, 34(23):5555-5565 doi: 10.1021/acs.organomet.5b00768
-
[27]
Wagner M, Zobel B, Dietz C, et al. Organometallics, 2015, 34(23):5602-5608 doi: 10.1021/acs.organomet.5b00829
-
[28]
Jímenez-Pérez V M, Camacho-Camacho C, Güizado-Rodríuez M, et al. J. Organomet. Chem., 2000, 614-615:283-293 doi: 10.1016/S0022-328X(00)00689-6
-
[29]
Hong M, Yin H, Chen S, et al. J. Organomet. Chem., 2010, 695(5):653-662 doi: 10.1016/j.jorganchem.2009.11.035
-
[30]
Williams D H, Fleming I, Translated by ZHANG Yan(张艳), QIU Di(邱頔), SHI Wei-Feng(施卫峰), et al. Spectroscopic Methods in Organic Chemistry. 6th Ed(有机化学中的光谱方法. 6版). Beijing: Peking University Press, 2015.
-
[31]
Effenberger K, Breyer S, Schobert R. Eur. J. Med. Chem., 2010, 45(5):1947-1954 doi: 10.1016/j.ejmech.2010.01.037
-
[32]
Dandawate P, Khan E, Padhye S, et al. Bioorg. Med. Chem. Lett., 2012, 22(9):3104-3108 doi: 10.1016/j.bmcl.2012.03.060
-
[33]
赵国良, 施霞, 张均平, 等.中国科学:化学, 2010, 40(10):1525-1535 http://www.cnki.com.cn/Article/CJFDTOTAL-SYQY201603027.htmZHAO Guo-Liang, SHI Xia, ZHANG Jun-Ping, et al. Sci. China:Chem., 2010, 40(10):1525-1535 http://www.cnki.com.cn/Article/CJFDTOTAL-SYQY201603027.htm
-
[34]
XU Jin-Gou(许金钩), WANG Zun-Ben(王尊本). Fluorescence Analysis. 3rd Ed(荧光分析法. 3版). Beijing: Science Press, 2006: 64-70
-
[35]
邵莺, 童晶晶, 吴祝宏, 等.常州大学学报:自然科学版, 2016, 28(6):38-43 http://www.cnki.com.cn/Article/CJFDTOTAL-SYQY201603027.htmSHAO Ying, TONG Jing-Jing, WU Zhu-Hong, et al. J. Changzhou Univ.:Nat. Sci. Ed., 2016, 28(6):38-43 http://www.cnki.com.cn/Article/CJFDTOTAL-SYQY201603027.htm
-
[36]
郑建华, 刘俊, 肖尧, 等.应用化学, 2015, 32(5):562-569 doi: 10.11944/j.issn.1000-0518.2015.05.140324ZHENG Jian-Hua, LIU Jun, XIAO Yao, et al. Chin. J. Appl. Chem., 2015, 32(5):562-569 doi: 10.11944/j.issn.1000-0518.2015.05.140324
-
[37]
赵国良, 沈金杯, 施霞, 等.无机化学学报, 2012, 28(5):959-964 http://www.wjhxxb.cn/wjhxxbcn/ch/reader/view_abstract.aspx?flag=1&file_no=20120517&journal_id=wjhxxbcnZHAO Guo-Liang, SHEN Jin-Bei, SHI Xia, et al. Chinese J. Inorg. Chem., 2012, 28(5):959-964 http://www.wjhxxb.cn/wjhxxbcn/ch/reader/view_abstract.aspx?flag=1&file_no=20120517&journal_id=wjhxxbcn
-
[38]
Yin H, Liu H, Hong M, et al. J. Organomet. Chem., 2012, 713(15):11-19
-
[1]
-
表 1 配合物的晶体学数据
Table 1. Crystallographic data of complexes C1 and C2
Complex C1 C2 Empirical formula C54H58N6O12Sn2 C31H38N2Sn Formula weight 1 220.44 621.32 T/K 296(2) 296(2) Crystal system Triclinic Monoclinic Space group P1 P21/n a/nm 1.074 53(7) 1.829 97(7) b/nm 1.130 91(8) 0.870 01(3) c/nm 1.301 50(9) 1.929 44(7) α/(°) 93.612 0(10) 90 β/(°) 100.471 0(10) 90.596 0(10) γ/(°) 116.373 0(10) 90 Volume/nm3 1.374 70(16) 3.071 68(19) Z 1 4 Dc/(Mg·m-3) 1.474 1.344 Absorption coefficient/mm-1 0.974 0.867 F(000) 620 1280 θ range/(°) 2.61~25.10 3.05~25.10 Limiting indices -12 ≤ h ≤ 12, -13 ≤ k ≤ 13, -15 ≤ l ≤ 15 -21 ≤ h ≤ 21, -10 ≤ k ≤ 10, -23 ≤ l ≤ 23 Reflection collected, unique 12 667, 4 870 (Rint=0.015 4) 38 638, 5 463 (Rint=0.044 7) Completeness 0.994 0.999 Max. and min. transmission 0.844 2 and 0.829 1 0.838 9 and 0.825 5 Data, restraint, parameter 4 870, 0, 342 5 463, 31, 354 Goodness-of-fit on F2 1.027 1.087 Final R indices [I>2σ(I)] R1=0.020 7, wR2=0.053 5 R1=0.040 8, wR2=0.110 8 R indices (all data) R1=0.023 0, wR2=0.054 6 R1=0.047 6, wR2=0.114 1 (Δρ)max, (Δρ)min/(e.nm-3) 397, -259 1 136, -456 表 2 配合物的部分键长和键角
Table 2. Selected bond lengths (nm) and bond angles (°) of the complexes
C1 Sn1-C19 0.214 2(2) Sn1-C11 0.214 4(2) Sn1-O1 0.216 66(13) Sn1-N1 0.223 82(16) Sn1-O2 0.234 20(14) Sn1-O6 0.242 26(17) C19-Sn1-C11 164.55(8) C19-Sn1-O1 96.42(7) C11-Sn1-O1 92.01(7) C19-Sn1-N1 96.71(8) C11-Sn1-N1 98.29(7) 01-Sn1-N1 70.56(6) C19-Sn1-O2 91.26(7) C11-Sn1-O2 90.52(7) 01-Sn1-O2 140.14(5) N1-Sn1-O2 69.71(5) C19-Sn1-O6 86.38(9) C11-Sn1-O6 82.83(8) 01-Sn1-O6 77.53(6) N1-Sn1-O6 148.08(6) 02-Sn1-O6 142.15(5) C2 Sn1-C23 0.213 9(4) Sn1-O1 0.214 1(2) Sn1-C15 0.214 6(4) Sn1-N1 0.223 0(3) Sn1-O2 0.232 2(2) Sn1-O4 0.239 4(3) C23-Sn1-O1 97.71(12) C23-Sn1-C15 164.08(16) 01-Sn1-C15 93.66(14) C23-Sn1-N1 97.43(16) 01-Sn1-N1 71.04(10) C15-Sn1-N1 96.78(13) C23-Sn1-O2 88.94(14) 01-Sn1-O2 141.09(9) C15-Sn1-O2 89.17(13) N1-Sn1-O2 70.09(9) C23-Sn1-O4 85.26(17) 01-Sn1-O4 78.98(10) C15-Sn1-O4 86.03(14) N1-Sn1-O4 150.00(10) 02-Sn1-O4 139.91(9) 表 3 配合物对癌细胞的体外抑制活性
Table 3. Inhibition action of complexes to cancer cell in vitro
Complex IC50/(μmol·L-1) H460 HepG2 MCF7 HL7702 C1 8.18±0.39 5.89±0.17 10.23±0.92 11.73±1.09 C2 4.18±0.13 1.89±0.55 2.41±0.51 6.61±0.33 Carboplatin 6.26±1.02 1.70±0.32 8.22±1.29 21.38±2.41 -

 扫一扫看文章
扫一扫看文章
计量
- PDF下载量: 0
- 文章访问数: 1823
- HTML全文浏览量: 237



 下载:
下载:







 下载:
下载:

