 图 1
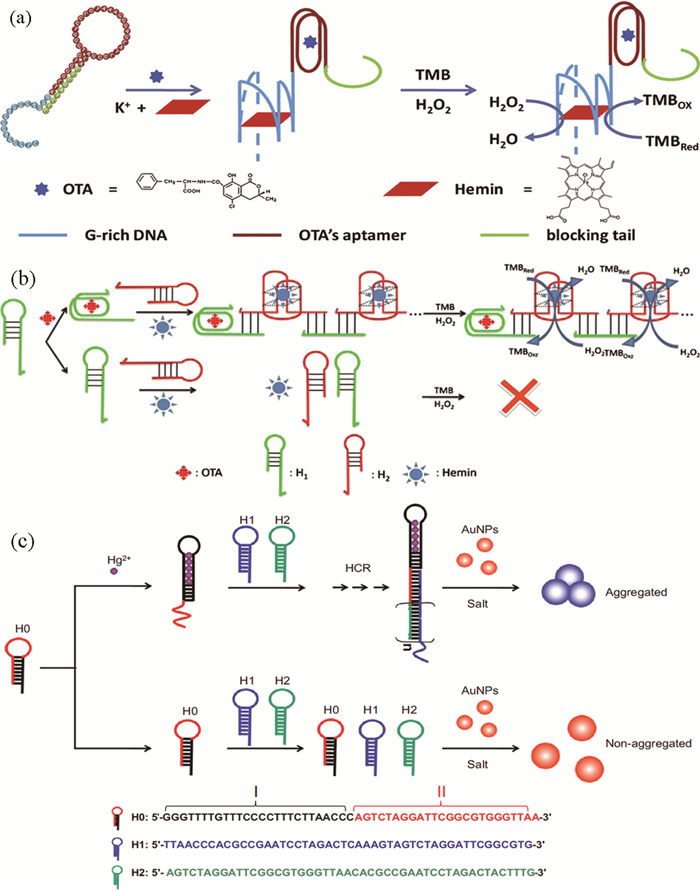
(a)基于“发夹”结构DNA[6]和(b)HCR比色法[7]检测赭曲霉毒素A(OTA)原理图,(c)基于HCR比色法检测Hg2+原理图[8]
Figure 1.
The illustrations of ochratoxin A detection using hairpin structure DNA (a) and HCR[7] (b), and the Hg2+ detecting process using HCR[8](c), respectively
图 1
(a)基于“发夹”结构DNA[6]和(b)HCR比色法[7]检测赭曲霉毒素A(OTA)原理图,(c)基于HCR比色法检测Hg2+原理图[8]
Figure 1.
The illustrations of ochratoxin A detection using hairpin structure DNA (a) and HCR[7] (b), and the Hg2+ detecting process using HCR[8](c), respectively

Citation: Wang Chengke, Wang Qingqing, Chen Dan. Research Progress in Hairpin Structure DNA used in the Biosensor[J]. Chemistry, 2017, 80(5): 420-427.

“发夹”结构DNA用于生物分析传感器研究进展
English
Research Progress in Hairpin Structure DNA used in the Biosensor
-
Key words:
- Hybridization chain reaction
- / DNA
- / Hairpin structure
- / Biosensor
-
DNA与人类生命密切相关,通过设计不同的碱基序列,可以使单链DNA发生链内碱基互补配对,自发形成首尾相连的“发夹”结构(见图 1(a)),并保持相对稳定的空间构型[1, 2]。“发夹”结构的DNA在一定条件下能够打开双链,并连续互补配对,形成更稳定的长链结构,称为杂交链式反应(Hybridization chain reaction,HCR)[3, 4]。由于DNA性质稳定,碱基序列易于调控,且易于修饰上其他的生物信号分子,在杂交链式反应中,由于每一个待测目标分子都可以触发一个HCR,同时产生一系列检测信号,使检测信号明显增强,因此,“发夹”结构的DNA在现代生物分析传感器的研制中受到人们的广泛关注,并得到广泛应用[5]。本文对其分类、信号记录方式及其在生物分子检测中的应用进行综述。
 图 1
(a)基于“发夹”结构DNA[6]和(b)HCR比色法[7]检测赭曲霉毒素A(OTA)原理图,(c)基于HCR比色法检测Hg2+原理图[8]
Figure 1.
The illustrations of ochratoxin A detection using hairpin structure DNA (a) and HCR[7] (b), and the Hg2+ detecting process using HCR[8](c), respectively
图 1
(a)基于“发夹”结构DNA[6]和(b)HCR比色法[7]检测赭曲霉毒素A(OTA)原理图,(c)基于HCR比色法检测Hg2+原理图[8]
Figure 1.
The illustrations of ochratoxin A detection using hairpin structure DNA (a) and HCR[7] (b), and the Hg2+ detecting process using HCR[8](c), respectively
1 基于“发夹”结构DNA的传感技术分类
依据分析传感器中涉及到的初始DNA的数目,可以将传感体系分为单链、双链和三链反应。
1.1 单链体系
当反应体系只涉及一条“发夹”结构的DNA单链时,如图 1(a)所示,DNA由三部分组成,分别为可识别靶标分子的适配体、信号分子单元(往往为具有催化能力的DNA酶片段)及对“发夹”结构起稳定作用的尾巴结构组成。在加入待测物时,待测物与其适配体特异性结合,封闭的“发夹”结构被打开,“发夹”结构上的DNA酶片段能够与氯化高铁血红素形成G-四联体结构,催化H2O2氧化四甲基联苯胺(TMB)显色,从而获得检测信号[6]。该方法无需使用抗体,DNA序列相对简单,信号可视化程度高,被广泛用于小分子的灵敏快速检测。
1.2 双链杂交链式反应
当反应体系涉及两条链时,如图 1(b)所示,可识别靶标物的适配体与DNA酶片段分别位于H1的5’端和H2的中部;同时,设计H1的3’端碱基与H2的3’端碱基互补,H2的5’端碱基与H1的5’端互补。加入待测物后,待测物与H1的5’端的适配体结合形成待测物与适配体的复合物,使H1的“发夹”结构打开,新暴露的3‘端与H2的3’端碱基互补,使H2的“发夹”结构打开,并暴露出H2的5’端,进一步与H1的5’端发生碱基互补反应,从而实现单一检测分子引发HCR并形成DNA长链。由于每一个H2分子中部的DNA酶片段均可以在血红素存在时引发H2O2氧化TMB显色,从而使检测信号得到级联放大,提高了检测的灵敏度[7]。
1.3 三链杂交链式反应
更为常见的是三条DNA链参与的HCR。如图 1(c)所示,靶标分子首先与DNA1(H0) 相互作用,诱导“发夹”结构的DNA1构型发生改变,使“发夹”结构打开,暴露出能与DNA2(H1) 相互作用的碱基序列,引发DNA2“发夹”结构打开,打开后的DNA2又可以与DNA3(H2) 循环相互作用,在DNA2和DNA3间发生HCR,形成长链,使检测信号大大增强。该方法的优点在于DNA1作为引发链,并不参与后续循环,降低了碱基序列的设计难度,使检测方案更加灵活、易于调控[8]。
2 检测信号记录方式
依据检测信号的记录方式,基于“发夹”结构DNA的传感技术主要有以下三种。
2.1 荧光法
在具有“发夹”结构的DNA上引入荧光基团或能够与荧光分子特异性结合的碱基序列,当体系中没有待测分子时,由于碱基互补配对作用,“发夹”结构的DNA双链呈闭合状态,荧光分子无法结合到DNA上或荧光信号被抑制;当体系中加入待测分子时,待测分子与“发夹”结构DNA相互作用,通过改变其空间构型,使双链打开,从而使荧光分子暴露或与荧光分子结合,使体系的荧光信号增强。利用“发夹”结构DNA参与的HCR,可以获得长链DNA,从而实现单一靶标分子诱导的多个荧光分子信号释放,使检测信号显著增强[9, 11]。荧光法具有背景低、检测限低、方法灵敏度高的优点,但往往会受限于所选取的荧光分子的性质以及荧光信号易“漂白”、不稳定的不足。荧光法主要通过引入荧光基团来达到信号放大的目的。用于功能化发夹结构DNA所使用荧光基团主要有NMM(N-methyl mesoporphyrin Ⅸ)、羧基荧光素(FAM)、SYBR Green Ⅰ等。
2.2 比色分析法
比色分析法具有检测可视化程度高、快速、灵敏、成本低、通用性强等优点,适合用于样品中微量成分的快速定性或半定量分析。不足在于检测对象相对单一,多集中于DNA,不适合同时检测复杂样品中的多种物质;当使用纳米金/银作为探针时,检测结果往往会受样品基质的影响,干扰物质较多。
2.3 电化学方法
电化学检测技术具有灵敏度高、操作简单、检测手段多样化(如电流、电压、阻抗、光电转换等)、检测成本低、耗样量少等优点,在生物工程、临床检验、基因诊断、药物筛选等方面有广阔的应用前景[27, 28]。近些年来,将“发夹”结构DNA应用于电化学分析的报道也层出不穷。通过在电极表面引发反应,形成长链DNA片段,可以改变电极表面结构与形貌,影响电极电子传输效率;当进一步引入电活性物质,如二茂铁[29]、亚甲基蓝[30]、Au/Ag纳米粒子[31, 32]、硫堇[33, 34]、石墨烯[35]等,可将电信号进一步增强,从而实现对靶标分子的电化学传感分析。该技术的不足在于电极修饰过程往往耗时较长,需要层层组装,电极再生步骤较繁琐,给实际应用带来不便。
2.1.2 采用FAM荧光基团
Li等根据聚多巴胺(PDA)纳米材料和HCR设计了非酶生物反应器检测DNA。发夹结构上标记FAM荧光分子,目标DNA促发HCR形成较长的双链,荧光信号明显增强。由于PDA功能化的Fe3O4纳米粒子与DNA粘性末端之间强的相互作用,利用磁铁分离作用,使溶液上清液中荧光分子数量减少,造成荧光信号显著下降[14]。
Ma等[15]结合适配体的特殊识别能力、氧化石墨烯猝灭荧光信号以及HCR的信号放大作用,设计了简单快速检测多肽的新方法。首先在发夹结构上修饰FAM荧光基团,存在待测物时,适配体与待测物结合暴露出引发片段,促发含有荧光分子的“发夹”结构DNA间HCR,形成DNA长链结构,由于空间构型改变带来的位阻效应,荧光分子远离氧化石墨烯,使体系具有很强的荧光信号。没有待测物时,荧光分子修饰的DNA由于π-π键共轭作用,以平铺形式结合到氧化石墨烯表面,荧光猝灭,如图 2(b)所示。
2.1.3 采用SYBR Green Ⅰ荧光基团
Dai等[16]利用抗体识别肿瘤坏死因子(Tumornecrosis factor,TNF),将另一个生物素化的抗体引入引发链DNA,通过HCR形成DNA长链,最后利用荧光分子SYBR Green Ⅰ特异性地结合到双链上,从而获得荧光检测信号。
Wang等[17]基于HCR对蛋白质进行检测。自由的发夹结构可以附着在石墨烯表面,靶标分子促发HCR形成长的双链DNA结构并脱离石墨烯表面。SYBR Green Ⅰ结合到双链DNA结构的缺口处,荧光信号增强。
2.1.1 采用NMM荧光基团
Chen等[12]设计了无酶免标记荧光生物传感器,三磷酸腺苷(ATP)适配体可以与包含DNA酶片段的单链DNA发生互补作用,从而抑制后者构型发生改变以及G-四联体的形成,进而抑制双链DNA与荧光分子NMM的结合。而当体系中加入ATP后,ATP与其适配体相互作用,引发DNA双链打开,解链后的DNA进一步与另两个“发夹”结构的DNA相互作用,引发HCR,形成包含一系列G-四联体结构的DNA长链,能够结合大量的NMM分子,从而使体系的荧光强度大大增强。基于此原理,能够实现对ATP的灵敏检测,检测线性范围为30~800 μmol/L,检测限为15μmol/L。
Guo等[13]利用HCR信号放大技术结合荧光法检测人类免疫缺陷病毒(HIV)DNA。利用荧光分子NMM结合到HCR形成的双链缺口处,使荧光信号显著增强。该方法可以在没有复杂仪器的情况下快速检测HIV DNA,且不需要标记和酶的参与,最低检测限达到0.5nmol/L。
2.2.1 基于金纳米颗粒聚集/分散的比色法
金纳米粒子(AuNPs)具有优异的光学性能,当分散状态的AuNPs在高浓度盐溶液(如NaCl)存在时会发生聚集,溶液颜色由红色转变为蓝紫色。AuNPs上存在长链DNA时,由于空间位阻效应,会抑制DNA的聚集[18, 19]。基于此,可以利用“发夹”结构DNA参与的HCR实现对靶标分子的比色传感分析。
Ma等[20]利用这一原理实现了对靶标DNA的灵敏检测。没有靶标DNA时,结合在AuNPs表面的DNA不能引发HCR。AuNPs在高浓度NaCl存在时发生聚集,靶标DNA一端可以与AuNPs表面DNA发生碱基互补配对作用,另一端可以引发HCR,使AuNPs表面的DNA长度增加,进而使AuNPs能够在高盐溶液中保持分散状态。利用简单的分光光度计或者裸眼观察,即可实现对靶标DNA的定性定量分析,如图 2(b)所示。
另一种思路是利用单链DNA或“发夹”结构DNA尾端带有大量负电荷,通过静电吸附作用可以结合到AuNPs表面,提高其在NaCl溶液中的稳定性,而靶标分子引发的HCR会促使大量的“发夹”结构DNA彼此互补配对,形成双链,从而消耗了溶液中游离的“发夹”结构DNA,减少了其对于AuNPs的保护作用,使AuNPs在NaCl溶液中发生聚集,溶液颜色由红色变为蓝紫色[8]。
Gao等[21]利用HCR信号放大的功能设计了利用比色法检测ATP的新方法。在此方法中,当ATP存在时,发夹结构打开促发HCR。AuNPs在高盐溶液中失去保护作用发生聚集,溶液颜色发生变化。
Liu等[22]利用AuNPs比色法和HCR信号放大功能设计了检测目标DNA的新方法,裸眼观察即可实现DNA的定量检测。该方法与之前基于AuNPs的检测方法相比,具有检测限低,特异性强的优点。
2.2.2 基于DNA酶比色法
Shimron等[23]利用G-四联体结构高催化活性的原理设计了检测目标DNA的新方法,当目标DNA乳腺癌易感基因(BRCA-1) 存在时,发夹结构打开并促发HCR。发夹结构上的酶片段暴露,在K+存在时,与血红素共同作用生成G-四联体结构。G-四联体结构具有辣根过氧化物酶(HRP)活性,催化H2O2氧化ABTS(2′-联氨-双-3-乙基苯并噻唑啉-6-磺酸)形成绿色产物,最低检测限为0.1pmol/L。
基于同样的原理,Dong等[24]结合G-四联体结构和HCR设计了检测双链DNA或RNA的新方法。G-四链体结构的序列三分之二封闭在发夹探针的环区,三分之一封闭在发夹探针的茎杆区。当目标靶分子存在时,发夹探针打开,G-四联体结构形成并催化ABTS显色。Xu等[25]首次利用了双茎发夹探针对与癌症相关的DNA序列进行检测,靶标分子存在时,促发剪切聚合循环释放G-四联体结构,G-四联体结构数量显著增加并催化ABTS显色。
Zhang等结合磁珠和HCR均具有放大信号的功能设计了可视化检测凝血酶的新方法。该方法设计了一段p-DNA与凝血酶适配体互补成双链DNA,利用生物素和亲和素之间的相互作用将双链DNA结合到磁珠表面,凝血酶与适配体片段特异性结合暴露出p-DNA片段并促发HCR形成较多G-四联体,当H2O2存在时催化TMB显色[26]。
2.3.3 基于碳材料的信号放大功能
Song等[35]结合了碳纳米管比表面积大的优点、HCR放大信号的功能和电化学免疫的灵敏性,设计了超灵敏、选择性强的电化学免疫传感器来检测EB病毒(EBNA-1)。最低检测限达到了0.7pg/mL,其检测方法稳定性好。
Liu等[38]利用三种信号协同放大作用检测乙型肝炎病毒基因组DNA,将二硫化钨-多壁碳纳米管复合材料和AuNPs修饰到电极表面,利用Au-S键相互作用在电极表面修饰cDNA。当靶标DNA存在时,cDNA与靶标DNA的3’末端反应固定靶标DNA,靶标DNA的5’末端可以打开标记有亲和素的发夹结构,促发HCR形成双链结构。利用生物素和亲和素之间的相互作用,标记有亲和素的HRP结合到双链结构上并催化氢醌产生电信号。
2.3.2 基于Au/Ag纳米材料的信号放大功能
Zhou等[31]首先在电极表面上引入俘获DNA(capture DNA),当溶液中存在靶标分子ATP时,由于ATP可以与溶液中的双链DNA上的适配体结合,促使其双链结构打开。解链后的DNA可以与capture DNA杂交,同时可以与溶液中另两条“发夹”结构的DNA发生HCR,从而在电极表面形成长链DNA片段。由于双链DNA可以与AgNO3作用,原位合成大量的Ag纳米材料,从而使电信号大大增强。基于此原理,可以实现对ATP的快速灵敏检测。
Li等[32]开发了一种超灵敏检测葡萄糖的电化学反应器。如图 2(c)所示,葡萄糖能够在葡萄糖氧化酶(GOD)和Fe2+存在时,引发DNA单链断裂,从而阻碍了电极表面HCR的进行,电极阻抗较大。而当体系中不含有葡萄糖时,DNA单链引发HCR进行,形成DNA长链,通过静电作用将带正电荷的AuNPs吸附到电极上,使电信号明显增强。同时利用HCR和AuNPs对电信号有双重放大作用,当存在葡萄糖时,引发HCR使电信号增大。基于此原理,能够对葡萄糖进行快速灵敏检测。该方法最低检测限为21pmol/L。
2.3.1 基于酶的信号放大功能
Zhao等[36]利用γ-干扰素与核酸适配体相互作用,减少了结合到电极表面适配体的数量,从而减少了含生物素分子的“发夹”结构DNA间HCR的发生,最后利用生物素与链霉亲和素的相互作用,将碱性磷酸酶固定到电极上,催化非电活性基团的萘磷酸转变为电活性的萘,利用差示脉冲伏安法实现了对γ-干扰素的灵敏检测。
Jia等[37]设计了超灵敏电化学生物反应器检测DNA,该检测方法特别之处是最后形成DNA分枝树。在此方法中引入了HCR,当加入目标DNA后,DNA片段根据碱基互补原则连续地结合到电极上,产生的DNA聚合物上带有大量暴露的HRP,最终使检测电流增大。
3 “发夹”结构DNA及杂交链式反应在分析传感中的应用
目前,由于其设计简单、信号放大效应明显、检测快捷灵敏的优点,“发夹”结构DNA的杂交链式反应被广泛应用于蛋白质[39~41]、DNA[42~45]、重金属离子[8, 46]、生物小分子[47, 48]、农药、真菌毒素[6, 7]等的检测。
3.1 蛋白质检测
蛋白质尤其是疾病蛋白及免疫球蛋白(IgG)的检测,对于疾病的早期发现和干预、疾病病理确定和研究、食品中过敏原的筛查等具有重要意义[49~51]。以往方法多采用基于抗原-抗体相互作用的酶联免疫法检测,往往需要价格昂贵的抗体、繁琐的实验步骤、耗时较长的检测周期,对操作人员的技术要求较高。更重要的是在不使用信号放大技术时,往往检测的灵敏度有限,不能满足临床上对疾病早发现、早治疗的要求[52, 53]。使用HCR及能特异性结合相应蛋白的核酸适配体,能更好地解决这一问题。
Song等[39]设计了基于适配体电化学传感器灵敏检测凝血酶的方法。在电极表面固定凝血酶的核酸适配体,从而可使复杂样品中的凝血酶特异性地结合到电极表面;再加入含有适配体和引发链的DNA时,引发链被结合到电极表面,通过引发链诱发含有生物素分子的两条DNA链发生HCR;最后,利用生物素与链霉亲和素特异性相互作用,将电活性的HRP大量结合到形成的长链DNA上,从而实现了电化学信号的显著增强,同时避免了使用抗体,降低了检测成本。
Xiao等[40]利用结合到电极表面的抗体识别IgG,再利用另一种生物素化的抗体在电极上引入生物素,进而利用链霉亲和素将含生物素分子的引发链连接到电极表面,最后引发链引发HCR,形成包含大量GOD的DNA长链,催化葡萄糖产生电信号。
3.2 DNA检测
DNA的灵敏检测对于临床检验、癌症基因的确定、遗传性疾病的早期筛查、干预和治疗以及转基因食品的检测都具有重要意义。
甲基化的DNA会促发HCR形成较长的双链,电信号增强;未甲基化的DNA在铜电极处被分解,不能引发HCR的进行,电信号无明显变化。Zhang等[42]基于此HCR信号放大原理实现了甲基化DNA的灵敏检测。
Chao等[43]开发了检测特定序列DNA的新方法,此方法使用荧光分子标记的发夹结构DNA,利用葡聚糖和HCR的协同作用放大信号。最低检测限达10fmol/L。
Yang等[54]开发了一种免酶电化学基因传感器检测乳腺癌基因BRCA-1。此传感器利用了HCR,比目前报道的使用酶或AuNPs的信号放大方法更灵敏,且选择性好,线性检测范围在1amol/L到10pmol/L。
3.3 重金属离子检测
近年来农产品与食品中重金属污染或重金属超标问题越来越引起人们的关注,农作物、食物和水中重金属超标对人体的伤害极大,因此对于重金属进行灵敏快速检测尤为重要。
Wang等[8]通过AuNPs比色方法及HCR信号放大技术,在Hg2+存在时,触发HCR生成长的双链DNA结构。双链不能有效地抑制AuNPs在高盐条件下的团聚,从而发展了一种检测Hg2+的新方法。
Chen等[46]基于能对Cu2+特异性响应的DNA酶(Cu2+-DNAzyme),通过荧光共振能量转移(FRET)技术开发了快速检测Cu2+的新方法。如图 2(a)所示,当Cu2+存在时,包含DNA酶的DNA结构被破坏,DNA引发链释放到溶液中,促发HCR形成带缺口的双链结构,在荧光分子FAM存在时发生FRET,实现了对Cu2+的快速灵敏检测。
3.4 其他小分子检测
小分子种类多,数量大。农兽药残留、人体内的营养物质、真菌毒素等都属于小分子范畴。由于与人类的生活息息相关,近年来小分子的检测引起了研究者的关注。
利用HCR放大信号的原理,Hun等[47]设计了一种化学免疫分析检测赤霉素的新方法。基于抗原和抗体的相互作用引入了能促发HCR的DNA片段。加入靶标分子时促发HCR,形成大量的富鸟嘌呤DNA双链结构并放大信号。
Feng等[9]设计了无酶灵敏检测腺苷的新方法。识别探针分为适配体1和适配体2两部分,适配体1和适配体2分别构成腺苷适配体的一部分。将适配体1固定到磁珠上起到富集和分离的作用,腺苷的识别作用可以使适配体1和溶液中的适配体2结合并形成了能够促发HCR的引发剂片段,此时将荧光分子SYBR Green Ⅰ结合到HCR形成的双链结构上。最终根据荧光信号变化对腺苷进行检测。
4 结语
DNA作为一种重要的生物分子,具有碱基序列可控、空间结构可调、易于人工合成、性质稳定、易于功能化的优点。基于“发夹”结构DNA的分析检测技术,受到人们越来越多的关注(见表 1)。尤其是HCR技术,能够实现信号多级放大、记录方式多样化,且往往可将信号分子和识别单元集成到DNA链内,无需使用抗体和大型检测仪器,从而大大降低检测成本,提高了检测的灵敏度、选择性和易操作程度,能够满足目前分析检测快速、灵敏、直观、经济、易操作的要求。
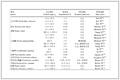 表 1
DNA“发夹”结构用于不同目标物的分析检测
Table 1.
Hairpin structure DNA used in the detection of different compounds.
表 1
DNA“发夹”结构用于不同目标物的分析检测
Table 1.
Hairpin structure DNA used in the detection of different compounds.

“发夹”结构DNA目前已经广泛应用于生物小分子、金属离子等的检测。今后,有望结合光、电、磁等检测技术,在研究细胞内DNA及蛋白相互作用、临床疾病标志物检测、疾病早期发现与治疗等方面有更为广泛的用途,从而实现节约检测成本和检测时间的目的。同时,基于“发夹”结构DNA的检测方法在制备小型化检测仪器以实现简便、快捷检测靶标分子方面也具有广阔的发展前景。
-
-
[1]
H Zhang, Y Yang, H L Dong et al. Biosens. Bioelectron., 2016, (86): 927~932.
-
[2]
B Wang, Y Y Wu, Y F Chen et al. Biosens. Bioelectron., 2016, (81): 125~130.
-
[3]
H L Shuai, K J Huang. L L Xing et al. Biosens. Bioelectron., 2016, (6): 337~345. https://www.researchgate.net/publication/304358462_Ultrasensitive_electrochemical_sensing_platform_for_MicroRNA_based_on_tungsten_oxide-graphene_composites_coupling_with_catalyzed_hairpin_assembly_target_recycling_and_enzyme_signal_amplification
-
[4]
H J Wang, W Jiang, Wei Li et al. Sens. Actuat. B, 2017, (238): 318~324. doi: 10.1080/10408347.2016.1176889?scroll=top&needAccess=true&journalCode=batc20
-
[5]
R M Dirks, N A Pierce. PNAS, 2004, (101): 15275~15278. https://www.researchgate.net/publication/220846098_Activatable_Tiles_Compact_Robust_Programmable_Assembly_and_Other_Applications
-
[6]
C Yang, V Lates, B P Simón et al. Biosens. Bioelectron., 2012, (32): 208~212. https://www.researchgate.net/publication/258034744_Rapid_high-throughput_analysis_of_Ochratoxin_A_by_the_self-assembly_of_DNAzyme-aptamer_conjugates_in_wine
-
[7]
C K Wang, X Y Dong, Q Liu et al. Anal. Chim. Acta, 2015, (860): 83~88. https://www.researchgate.net/publication/272296676_Label-free_colorimetric_aptasensor_for_sensitive_detection_of_ochratoxin_A_utilizing_hybridization_chain_reaction
-
[8]
Q Wang, X H Yang, P Liu et al. Spectrochim. Acta A, 2015, (136): 283~287. https://www.researchgate.net/publication/269169672_Colorimetric_detection_of_mercury_ion_based_on_unmodified_gold_nanoparticles_and_target-triggered_hybridization_chain_reaction_amplification?ev=auth_pub
-
[9]
C J Feng, Z Hou, J W et al. Biosens. Bioelectron., 2016, (86): 966~970. https://www.researchgate.net/publication/229299462_A_study_on_a_novel_superoxide_dismutase_mimic_bisdioxotetraamine_copperII_complex_-_Kinetics_of_catalytic_dismutation_of_superoxide_anion_by_pulse_radiolysis_and_solution_equilibrium?citationList=outgoi
-
[10]
Y L Wei, W J Zhou, X Li et al. Biosens. Bioelectron., 2016, (77): 416~420.
-
[11]
X X Lv, W C Wu, C G Niu et al. Talanta, 2016, (151): 62~67. https://www.researchgate.net/publication/290479608_A_facile_turn-on_fluorescent_method_with_high_sensitivity_for_Hg2_detection_using_magnetic_Fe3O4_nanoparticles_and_hybridization_chain_reactions?ev=auth_pub
-
[12]
Q G Chen, Q Q Guo, Y Chen et al. Talanta, 2015, (138): 15~19. https://www.researchgate.net/publication/272364830_An_enzyme-free_and_label-free_fluorescent_biosensor_for_small_molecules_by_G-quadruplex_based_hybridization_chain_reaction
-
[13]
Q Q Guo, Y Chen, Z P Song et al. Anal. Chim. Acta, 2014, (852): 244~249. http://www.researchgate.net/publication/268752582_Label-free_and_enzyme-free_sensitive_fluorescent_detection_of_human_immunodeficiency_virus_deoxyribonucleic_acid_based_on_hybridization_chain_reaction?ev=auth_pub
-
[14]
N Li, X Hao, B H Kang et al. Biosens. Bioelectron., 2016, (77): 525~529.
-
[15]
C Ma, H Y Liu, T Tian et al. Biosens. Bioelectron., 2016, (83): 15~18. https://www.researchgate.net/publication/301275410_A_simple_and_rapid_detection_assay_for_peptides_based_on_the_specific_recognition_of_aptamer_and_signal_amplification_of_hybridization_chain_reaction
-
[16]
S Dai, C J Feng, W Li et al. Biosens. Bioelectron., 2014, (60): 180~184. https://www.researchgate.net/publication/260841914_Plasma_microRNA_Expression_and_Micronuclei_Frequency_in_Workers_Exposed_to_Polycyclic_Aromatic_Hydrocarbons
-
[17]
X Z Wang, A W Jiang, T Hou et al. Biosens. Bioelectron., 2015, (70): 324~329. https://www.researchgate.net/publication/274394316_Enzyme-free_and_label-free_fluorescence_aptasensing_strategy_for_highly_sensitive_detection_of_protein_based_on_target-triggered_hybridization_chain_reaction_amplification
-
[18]
A Nazirov, A Pestov, Y Privar et al. Carbohydr. Polym., 2016, (151): 649~655. https://www.researchgate.net/publication/303815707_One-pot_Green_Synthesis_of_Luminescent_Gold_Nanoparticles_Using_Imidazole_Derivative_of_Chitosan
-
[19]
R Ionescu, U Cindemir, T Geremariam et al. Sens. Actuat., B, 2017, (239): 455~461. http://uu.diva-portal.org/smash/record.jsf?pid=diva2%3A958089&dswid=-1663
-
[20]
C P Ma, W S Wang, A Mulchandani et al. Anal. Biochem., 2014, (457): 19~23. https://www.researchgate.net/publication/262341606_A_simple_colorimetric_DNA_detection_by_target-induced_hybridization_chain_reaction_for_isothermal_signal_amplification
-
[21]
Z Gao, Z L Qiu, M H Lu et al. Biosens. Bioelectron., 2017, (89):1006~1012. https://www.researchgate.net/publication/49629065_PAK1IP1_a_ribosomal_stress-induced_nucleolar_protein_regulates_cell_proliferation_via_the_p53-MDM2_loop
-
[22]
P Liu, X H Yang, S Sun et al. Anal. Chem., 2013, (85): 7689~7695. https://www.researchgate.net/publication/253337162_Enzyme-Free_Colorimetric_Detection_of_DNA_by_Using_Gold_Nanoparticles_and_Hybridization_Chain_Reaction_Amplification
-
[23]
S Shimron, F Wang, R Orbach et al. Anal. Chem., 2012, (84): 1042~1048. https://www.researchgate.net/publication/221745401_Amplified_Detection_of_DNA_through_the_Enzyme-Free_Autonomous_Assembly_of_HeminG-Quadruplex_DNAzyme_Nanowires
-
[24]
J Dong, X Cui, Y Deng et al. Biosens. Bioelectron., 2012, (38): 258~263. https://www.researchgate.net/publication/228081654_Amplified_detection_of_nucleic_acid_by_G-quadruplex_based_hybridization_chain_reaction
-
[25]
J G Xu, H L Li, Z S Wu et al. Theranostics. 2016, (6): 318~327. https://www.researchgate.net/publication/288903841_Double-stem_Hairpin_Probe_and_Ultrasensitive_Colorimetric_Detection_of_Cancer-related_Nucleic_Acids
-
[26]
Y Zhang, W Ren, H Q Luo et al. Biosens. Bioelectron. 2016, (80): 463~470. https://www.researchgate.net/publication/293642191_Label-free_cascade_amplification_strategy_for_sensitive_visual_detection_of_thrombin_based_on_target-triggered_hybridization_chain_reaction-mediated_in_situ_generation_of_DNAzymes_and_Pt_nanochains
-
[27]
Y H Wang, L Jiang, L Chu et al. Biosens. Bioelectron., 2017, (87): 459~465. https://www.researchgate.net/publication/307627972_Electrochemical_detection_of_glutathione_by_using_thymine-rich_DNA-gated_switch_functionalized_mesoporous_silica_nanoparticles
-
[28]
Y Lv, L L Yang, X X Mao et al. Biosens. Bioelectron., 2016, (85): 664~668. https://www.researchgate.net/publication/303511414_Electrochemical_detection_of_glutathione_based_on_Hg2-mediated_strand_displacement_reaction_strategy
-
[29]
Z B Li, X M Miao, K Xing et al. Biosens. Bioelectron., 2015, (74): 687~690. https://www.researchgate.net/publication/280327255_Enhanced_electrochemical_recognition_of_double-stranded_DNA_by_using_hybridization_chain_reaction_and_positively_charged_gold_nanoparticles
-
[30]
彭德敏, 袁若. 化学传感器, 2014, 34: 15~19. http://kns.cnki.net/KCMS/detail/detail.aspx?filename=hxch201404002&dbname=CJFD&dbcode=CJFQ
-
[31]
Q Zhou, Y X Lin, Y P Lin et al. Talanta, 2016, (146): 23~28. https://www.researchgate.net/publication/282997560_In_situ_amplified_electrochemical_aptasensing_for_sensitive_detection_of_adenosine_triphosphate_by_coupling_target-induced_hybridization_chain_reaction_with_the_assembly_of_silver_nanotags
-
[32]
Z B Li, X M Miao, A H Zhu et al. Biochem. Eng. J., 2015, (103): 205~210. https://www.researchgate.net/publication/282794537_Hybridization_chain_reaction_and_gold_nanoparticles_dual_signal_amplification_for_sensitive_glucose_detection
-
[33]
Z Zhang, L Q Luo, L M Zhu et al. Analyst, 2013, (138): 5365~5370.
-
[34]
L M Zhu, L Q Luo, Z X Wang et al. Biosens. Bioelectron., 2012, (35): 507~511. https://www.researchgate.net/publication/223965636_DNA_electrochemical_biosensor_based_on_thionine-graphene_nanocomposite
-
[35]
C Song, G M Xie, L Wang et al. Biosens. Bioelectron., 2014, (58): 68~74. https://www.researchgate.net/publication/260805101_DNA-based_hybridization_chain_reaction_for_an_ultrasensitive_cancer_marker_EBNA-1_electrochemical_immunosensor
-
[36]
J J Zhao, C F Chen, L L Zhang et al. Biosens. Bioelectron., 2012, (36): 129~134. https://www.researchgate.net/publication/224938947_An_electrochemical_aptasensor_based_on_hybridization_chain_reaction_with_enzyme-signal_amplification_for_interferon-gamma_detection
-
[37]
L P Jia, S S Shi, R N Ma et al. Biosens. Bioelectron., 2016, (80): 392~397. https://www.researchgate.net/publication/292951021_Highly_sensitive_electrochemical_biosensor_based_on_nonlinear_hybridization_chain_reaction_for_DNA_detection
-
[38]
X Liu, H L Shuai, Y J Liu et al. Sens. Actuat. B, 2016, (235): 603~613. https://www.researchgate.net/publication/303600665_An_electrochemical_biosensor_for_DNA_detection_based_on_tungsten_disulfidemulti-walled_carbon_nanotube_composites_and_hybridization_chain_reaction_amplification
-
[39]
W L Song, X X Xie, W B Sun et al. Anal. Chim. Acta, 2015, (860): 77~82. https://www.researchgate.net/publication/255736784_Clinical_analysis_and_prognostic_significance_of_hepatitis_B_virus_infections_for_diffuse_large_B-cell_lymphoma_with_or_without_rituximab_therapy
-
[40]
L J Xiao, Y Q Chai, R Yuan et al. Talanta, 2013, (115): 577~582. https://www.researchgate.net/publication/256859421_Amplified_electrochemiluminescence_of_luminol_based_on_hybridization_chain_reaction_and_in_situ_generate_co-reactant_for_highly_sensitive_immunoassay
-
[41]
J J Guo, J C Wang, J J Zhang et al.Biosens. Bioelectron., 2017, (90): 159~165. https://www.researchgate.net/publication/272193553_Porcine_antilymphocyte_globulin_p-ALG_plus_cyclosporine_A_CsA_treatment_in_acquired_severe_aplastic_anemia_a_retrospective_multicenter_analysis
-
[42]
L Q Zhang, Y J Liu, Y Li et al. Anal. Chim. Acta, 2016, (933): 75~81.
-
[43]
J Chao, Z Li, J Li et al. Biosens. Bioelectron., 2016, (81): 92~96. https://www.researchgate.net/publication/295396577_Hybridization_Chain_Reaction_Amplification_for_Highly_Sensitive_Fluorescence_Detection_of_DNA_with_Dextran_Coated_Microarrays
-
[44]
L Song, Y Zhang, J Li et al. Appl. Spectrosc., 2017, (70): 688~694.
-
[45]
X Ding, W Cheng, Y Li et al. Biosens. Bioelectron., 2017, (87): 345~351. https://www.researchgate.net/publication/229387076_Development_of_multi-gap_resistive_plate_chambers_with_low-resistive_silicate_glass_electrodes_for_operation_at_high_particle_fluxes_and_large_transported_charges
-
[46]
Y Chen, L Chen, Y D Ou et al. Talanta, 2016, (552): 245~249.
-
[47]
X Hun, Y Q Xu, C Chen et al. Sens. Actuat. B, 2014, (202): 594~599. https://www.researchgate.net/publication/270319281_DNA-based_hybridization_chain_reaction_for_signal_amplification_and_ultrasensitive_chemiluminescence_detection_of_gibberellic_acid
-
[48]
W T Xu, J J Tian, X L Shao et al. Biosens. Bioelectron., 2017, (89):795~801. https://www.researchgate.net/publication/308960805_A_rapid_and_visual_aptasensor_for_Lipopolysaccharides_detection_based_on_the_bulb-like_triplex_turn-on_switch_coupled_with_HCR-HRP_nanostructures
-
[49]
Y Huang, H K Huang, J Qin et al. Sens. Actuat. B, 2017, (238): 908~916.
-
[50]
S M Fang, X D Dong, S L Liu et al. Electrochim. Acta, 2016, (212): 1~9. https://www.infona.pl/resource/bwmeta1.element.elsevier-d0dc017a-fea1-3077-b1b1-963046ab60c2
-
[51]
N X Li, J Y Chen, M Luo et al. Biosens. Bioelectron., 2017, (87): 325~331. https://www.researchgate.net/publication/259698643_Highly_sensitive_and_multiple_DNA_biosensor_based_on_isothermal_strand-displacement_polymerase_reaction_and_functionalized_magnetic_microparticles?ev=auth_pub
-
[52]
M Sai, X M Ou, D K Zhu et al. J. Virol. Methods, 2016, (237): 79~85. https://www.researchgate.net/publication/307559139_Development_and_evaluation_of_indirect_ELISAs_for_the_detection_of_IgG_IgM_and_IgA1_against_duck_hepatitis_A_virus_1
-
[53]
O Hohn, S Mostafa, S Norley et al. J. Virol. Methods, 2016, (234): 186~192. https://www.researchgate.net/publication/301774519_Development_of_an_antigen-capture_ELISA_for_the_detection_of_the_p27-CAprotein_of_HERV-KHML-2
-
[54]
H Yang, Y Gao, S Q Wang et al. Biosens. Bioelectron., 2016, (80): 450~455. https://www.researchgate.net/publication/292950769_Mercury_mass_flow_in_iron_and_steel_production_process_and_its_implications_for_mercury_emission_control
-
[55]
D W Yang, L M Ning, T Gao et al. Electrochem. Commun., 2015, (58): 33~36. https://www.researchgate.net/publication/262056820_Decreased_miR-146_Expression_in_Peripheral_Blood_Mononuclear_Cell_Correlated_with_Ongoing_islet_autoimmunity_in_Type_1_Diabetes_Patients
-
[56]
Q Zhai, Y He, X L Li et al. J. Electroanal. Chem., 2015, (758): 20~25. https://www.researchgate.net/publication/283813657_A_simple_and_ultrasensitive_electrochemical_biosensor_for_detection_of_microRNA_based_on_hybridization_chain_reaction_amplification
-
[57]
Y Qian, C Y Wang, F L Gao. et al. Biosens. Bioelectron., 2015, (63):425~431. https://www.researchgate.net/publication/264830561_Ultrasensitive_electrochemical_detection_of_DNA_based_on_Zn2_assistant_DNA_recycling_followed_with_hybridization_chain_reaction_dual_amplification
-
[58]
Y H Wang, L Jiang, Q G Leng et al. Biosens. Bioelectron., 2016, (74): 914~920. https://www.researchgate.net/publication/283494239_Electrochemical_Sensor_for_Glutathione_Detection_Based_on_Mercury_Ion_Triggered_Hybridization_Chain_Reaction_Signal_Amplification
-
[59]
Y Y Yu, Z G Chen, W S Jian et al. Biosens. Bioelectron., 2015, (64): 566~571. https://www.researchgate.net/publication/266947367_Ultrasensitive_electrochemical_detection_of_avian_influenza_A_H7N9_virus_DNA_based_on_isothermal_exponential_amplification_coupled_with_hybridization_chain_reaction_of_DNAzyme_nanowires
-
[1]
-
表 1 DNA“发夹”结构用于不同目标物的分析检测
Table 1. Hairpin structure DNA used in the detection of different compounds.

-

 扫一扫看文章
扫一扫看文章
计量
- PDF下载量: 0
- 文章访问数: 0
- HTML全文浏览量: 0


 下载:
下载:

 下载:
下载:

