
Citation: ZHAO Teng-Teng, YANG Xue-Yu, DONG Ke-Ke, ZHU Xiao-Lei. Interaction Mechanism Between AChE and 1, 7-Diazacarbazole Inhibitors Based on Molecular Dynamics Simulations[J]. Chinese Journal of Inorganic Chemistry, 2017, 33(11): 2065-2074. doi: 10.11862/CJIC.2017.250

乙酰胆碱酯酶AChE与1,7-二氮杂咔唑抑制剂的作用机理的分子动力学模拟
-
关键词:
- 阿尔茨海默症
- / 乙酰胆碱酯酶
- / 1,7-二氮杂咔唑类抑制剂
- / 分子动力学模拟
- / MM-PBSA
English
Interaction Mechanism Between AChE and 1, 7-Diazacarbazole Inhibitors Based on Molecular Dynamics Simulations
-
Key words:
- Alzheimer's disease
- / AChE
- / 1, 7-diazacarbazole inhibitor
- / molecular dynamics simulation
- / MM-PBSA
-
众所周知,世界正面临老龄化问题,阿尔茨海默病(AD)的发病率随着年龄的增加而显著增加。阿尔茨海默症病人的主要表现为认知功能障碍[1]、记忆缺失及语言障碍[2-3]等神经精神症状。迄今为止,阿尔茨海默病本质上可能的病因是基底前脑胆碱能神经递质的缺陷,尤其是乙酰胆碱(ACh)的大量缺失[4-6]。胆碱酯酶包括乙酰胆碱酯酶(AChE)和丁酰胆碱酯酶(BChE),其主要作用是使胆碱能神经递质水解。在ACh的水解酶作用下[7],ACh将被水解为胆碱和乙酸,从而引起阿尔茨海默症。因此,抑制AChE和BChE被认为是最有前途的治疗阿尔茨海默病的策略。
根据X射线结构分析[8],AChE的活性位点中存在一个狭而深的峡谷,包括两个不同的结合位点:位于峡谷底部的Ser-His-Glu催化位点(CAS),以及位于峡谷入口的外围阴离子结合位点(PAS)[8]。因此,AChE抑制剂可分为两种类型,即单位点抑制剂和双位点抑制剂[8-12]。前人研究[10, 13]表明,与单位点抑制剂相比,双位点抑制剂能更有效的治疗AD。
至今,实验上已经设计合成了许多AChE和BChE抑制剂。例如,Xie等[14]设计合成了一系列新型的具有多功能多靶点的香豆素类混合抑制剂,其中大多数抑制剂能有效地抑制AChE的活性。Alpan等[15]设计合成了苯并咪唑及其衍生物类抑制剂,并进行了活性分析,发现改变杂环上的亲脂性原子或者环上的取代基均可增强AChE抑制剂的活性。Maryam等[16]合成了一系列的新型的吖啶酮连接到1,2,3,-三唑的衍生物,并预测了它们对AChE和BChE的生物活性,对接结果与实验结果相一致。Asadipour等[17]设计合成了一系列由香豆素、甲酰胺基以及苄基哌啶这几个结构组成的苄基哌啶类AChE抑制剂,对接研究表明它们具有双重结合位点。Gao等[18]和Liu等[19]设计合成了一系列新型的氯查耳酮的叔胺衍生物,并评估了这一类衍生物对AChE和BChE的生物活性。最近,Gazzard等[20]发现,1,7-二氮杂咔唑衍生物是AChE强有效的抑制剂。在理论研究方面,Niu等[21]采用拉伸分子动力学模拟方法研究了抑制剂E2020与AChE的结合机制,并在原子水平上为E2020从乙酰胆碱酯酶(TcAChE)的解离提供了新的思路。Puiatti等[22]通过分子对接及分子动力学模拟对抑制剂与AChE的结合模式进行了研究,并比较了3种石松类生物碱抑制剂与AChE的结合模式,通过MM-GBSA方法计算的3个复合物的结合能与实验上抑制剂的IC50值相一致。Zhu等[23]利用分子动力学和MM-PBSA方法,研究了syn1-TZ2PA6和anti1-TZ2PA6两种同分异构体分别对AChE1和AChE2的选择性机制。Zhou等[24]通过3D-QSAR方法、分子对接和分子动力学模拟对60种他克林衍生物及其对AChE抑制活性的影响进行了研究。结果表明,AChE与抑制剂形成的复合物的空间位阻和氢键对抑制剂的生物活性影响较大。AChE的结合位点的关键残基包含Tyr70、Trp84、Tyr121、Trp279和Phe330。Shi等[25]用分子对接和分子动力学模拟方法,研究了对映体(RS,S)-17b和(RS,R)-17b与AChE的结合模式,为设计新型高效的AChE抑制剂提供了新的线索。最近,Mohammadi等[26]通过改变烷基取代基设计出了许多氨基甲酸酯衍生物,并利用分子对接和分子动力学模拟方法研究了取代结构对抑制剂与AChE结合能以及AChE构象变化的影响。Hossain等[27]在化学计量学和动力学模拟分析的基础上研究了乙酰胆碱酯酶抑制剂对分子结构的要求。
目前许多AChE抑制剂已经在临床上应用于AD的治疗,例如他克林(Tacrine)[28]、多奈哌齐(Donepezil)[29-30]、加兰他敏(Galanthamine)[31]、利凡斯的明(Rivastigmine)[32-33]等药物,但是治疗中仍然伴随着一定的副作用。上述问题出现的主要原因之一是抑制剂与AChE之间的作用机理还没有清楚地理解,这是一个亟待解决的问题。因此,设计高效的最小副作用的新型AChE抑制剂仍然是非常重要且具有挑战性的任务。目前,从原子层次上揭示抑制剂与AChE详细的作用机理还没有报道,而这些研究的进行有利于解决上述问题以及加速实验研究。在这个过程中,分子动力学模拟是一个关键性的技术[21-27]。因此,我们选择了3个1,7-二氮杂咔唑衍生物作为AChE抑制剂,利用分子对接、分子动力学模拟、自由能计算以及能量分解等方法来研究抑制剂与AChE的结合机理,揭示AChE抑制剂的生物活性大小的原因。模拟结果证明了范德华相互作用(vdW)对结合自由能有较大的贡献,静电相互作用(包括氢键作用)有利于3个抑制剂稳定在AChE的结合位点中。结合位点上的主要关键残基(W279和Y334),在抑制剂与AChE的结合中以及区分3个抑制剂的生物活性上发挥着重要作用。本文的研究为今后设计更高效的AChE抑制剂提供了理论指导。
1 实验部分
1.1 准备初始结构
AChE的X射线晶体结构是从PDB数据库(PDB代码:1EVE,分辨率为0.25 nm)[34]中提取的,去除该晶体结构中的配体、结晶水分子和氢原子,作为初始的蛋白质结构。利用画图软件构建3个1,7-二氮杂咔唑衍生物(分别标记为M1、M2和M3),如表 1中所示,并在B3LYP/6-31G*水平上用Gaussian 09软件[35]进行优化得到最优结构,优化后的结构作为分子对接所需配体的初始结构。
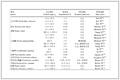 表 1
3个1, 7-二氮杂咔唑衍生物抑制剂的结构和IC50值
Table 1.
Structures and bioactivity of three 1, 7-diazacarbazole derivative inhibitors to AChE
表 1
3个1, 7-二氮杂咔唑衍生物抑制剂的结构和IC50值
Table 1.
Structures and bioactivity of three 1, 7-diazacarbazole derivative inhibitors to AChE

Inhibitor R IC50 / (nmol·mL-1) M1 
0.890 M2 
4.77 M3 
13.3 1.2 对接方法
小分子配体与受体的相互作用首先通过分子对接研究。我们采用Autodock4.0软件的拉马克遗传算法[36]进行了分子对接的研究,将极性氢原子添加到蛋白质的各个残基上,再将科尔曼联合原子电荷添加到AChE的部分原子上。在对接过程中,将受体的格点盒子大小设置为6.5 nm×6.5 nm×6.5 nm,间隔为0.037 5 nm。用遗传算法共进行200次独立对接计算,然后从对接构象的最大簇(均方根偏差RMSD设定为0.2 nm)选出最优构象,最终得到的复合物体系的结构用作后续的MD模拟的初始结构。
1.3 分子动力学模拟
我们采用AMBER10.0软件分别对3个复合物体系的进行MD模拟,本文中受体和配体的力场分别为AMBERFF03[37]和GAFF[38]力场,用Antechamber模块添加配体的氢原子并拟合RESP(限制静电势)[39]电荷。将3个复合物体系分别加到一个溶剂为TIP3P水分子模型[40]的盒子中,盒子半径为1.0 nm。氢原子通过LEaP模块添加,并添加抗衡离子Na+使体系电荷呈中性。在MD之前,对每个体系进行能量最小化,采用2 000步的最小化法和2 000步的共轭梯度法,非键截断半径设为1.0 nm。MD模拟过程先进行200 ps的41 840 kJ·mol-1·nm-2限制性加热(0~300 K),然后在NPT系综(P=101 kPa,T=300 K)下进行时间步长为0.002 ps的50 ns非限制的MD模拟。在MD模拟过程中,采用SHAKE算法[41]对原子键长进行约束,长程静电相互作用采用Particle-mesh Ewald (PME)[42]进行处理。
1.4 结合自由能计算
为了清楚地了解3个抑制剂与AChE的结合机制,我们通过AMBER10.0中的MM-PBSA程序[43]计算得到每个复合物体系的结合自由能以及能量分解。对于每个体系,取稳定后的最后5 ns的MD轨迹,提取200个构象对抑制剂与AChE的结合自由能计算来研究生物活性。配体与受体结合自由能(ΔGbind)可表述为[44-45]:
其中ΔGcomplex、ΔGprotein和ΔGligand分别代表复合物、蛋白质以及配体的自由能。结合自由能又可分解为三部分:气相结合能(ΔEgas)、溶剂化自由能(ΔGsol)和熵贡献(-TΔS)(式2)。ΔEgas又可进一步分解成静电能(ΔEele)和范德华作用能(ΔEvdW)(式3)。溶剂化自由能(ΔGsol)可以分为两部分:极性溶剂化自由能(ΔGpolar)和非极性溶剂化自由能(ΔGnonpolar)(式4)。非极性溶剂化自由能(ΔGnonpolar)通过计算方程(式5)中的溶剂可及表面积(SASA)获得,其中SASA可以通过AMBER 10.0软件中的Molsurf方法得到。溶剂的探针半径设为0.14 nm。对应的溶剂参数γ和β分别设为0.226 77 kJ· mol-1·nm-1和3.85 kJ·mol-1。溶质和溶剂的介电常数分别设为1.0和80.0。另外,由于熵的计算对于大体系而言异常耗时,并且对于相似的蛋白质来说小分子的结合引起的熵变很接近[46]。因此,在本文的工作中忽略了熵(-TΔS)对ΔGbind的贡献[46-47]。用这种方法对能量的计算在前人的研究工作中也被证实是合理的[48]。
2 结果与讨论
2.1 结合模式
在本文中,我们分别将抑制剂M1、M2和M3对接到AChE中,得到最优的对接构象如图 1(a)所示,将用于后续的MD模拟。从图 1(a)中可以看出,3个抑制剂与AChE的结合模式大致相同,详细的对接模式如图 2所示。在图 2中可以看出,所有抑制剂都位于由残基Y70(Tyr70)、V71(Val7l)、D72(Asp72)、S81(Ser81)、W84(Trp84)、N85(Asn85)、G118(Gly118)、Y121(Tyr121)、S122(Ser122)、W279(Trp279)、L282(Leu282)、S286(Ser286)、I287(Ile287)、F288(Phe288)、R289(Arg289)、F290(Phe290)、F330(Phe330)、F331(Phe331)、Y334(Tyr334)和W432(Trp432)形成的结合口袋中。同时,我们还可以看出3个抑制剂均与AChE形成了氢键,使抑制剂与AChE结合模式更加牢固。选择结合能较低且构象较多的结构作为最好的对接构象,用作后续MD模拟的初始结构。如图 1(b)和图 3所示,在MD模拟之后,3个复合物体系的构象与图 1(a)和图 2中的对接结果很相似,配体周围的残基变化不大。
2.2 MD过程中体系的稳定性和柔性分析
为了考察3个复合体的稳定性,我们分析了MD模拟过程中的结合态AChE上的骨架Cα原子的均方根偏差(RMSD)(图 4(a))。从图 4(a)中可以看出,3个体系的RMSD值均在15 ns之后趋于稳定,其值在0.15 nm附近波动,这表明这些复合物已经达到稳定。基于上述分析,我们取最后5 ns的模拟用于后续的构象和能量分析是合理的。另一方面,回旋半径(Rg)是一种研究药物对蛋白质尺寸影响的指标[49]。如图 4(b)所示,是3个结合态AChE的回旋半径随时间的变化曲线。AChE/抑制剂体系的Rg平均值大概为2.28~2.32 nm,每个体系的回旋半径基本上都能保持稳定。因此,抑制剂与AChE结合时,AChE的结构都是稳定的。
为了深入了解结合态AChE残基的变化,我们对蛋白质的柔性进行了分析,蛋白质的柔性可由每一个残基的B因子(B-factor)来评判。本文我们计算了3个复合物体系的蛋白质残基的B-factor值,并与游离态AChE的B-factor进行比较。通过分析,3个复合物体系具有相似的B-factor值分布,并且结合态AChE的残基与游离态AChE实验晶体结构的B-factor值分布基本一致,这也说明3个抑制剂与AChE的结合模式比较相似。
2.3 AChE与抑制剂的结合机理及抑制剂的生物活性
抑制剂与AChE的结合自由能计算是衡量抑制剂活性的重要方法。本文我们用MM-PBSA的方法计算了3个复合物体系的结合自由能以及不同能量项,结果列在表 2中。表 2中的能量值,正数表示这种作用不利于抑制剂与AChE的结合,负数表示这种作用使体系能量降低,有利于结合,负数绝对值越大,表示这种作用对结合的贡献越大。从表 2可以看出,M1、M2、M3与AChE的结合自由能分别为:-144.35、-129.37、-102.47 kJ·mol-1,与实验中抑制剂生物活性(IC50值分别为0.890、4.77、13.3 nmol·ml-1)[20]的大小次序相一致,表明抑制剂的生物活性顺序:M1>M2>M3。
 表 2
通过MM-PBSA方法计算得到的复合物的结合自由能
Table 2.
Binding free energy components for the proteininhibitor complexes by using the MM-PBSA methoda
表 2
通过MM-PBSA方法计算得到的复合物的结合自由能
Table 2.
Binding free energy components for the proteininhibitor complexes by using the MM-PBSA methoda
Componentb AChE/M1(SDc) AChE/M2(SD) AChE/M3(SD) ΔEele -56.48(15.65) -22.51(13.22) -27.41(19.41) ΔEvdw -241.17(9.33) -234.72(9.46) -200.58(10.04) ΔEgas -297.65(16.57) -257.23(13.93) -227.99(25.40) ΔEpol 181.42(13.77) 155.14(19.71) 152.47(28.62) ΔGnonpolar -28.16(0.63) -27.32(0.59) -26.99(0.88) ΔGsol 153.26(13.64) 127.86(19.87) 125.52(28.53) ΔGbind -144.35(15.02) -129.37(19.08) -102.47(19.00) IC50 0.890 4.77 13.3 aAll values are given in kJ·mol-1; bComponents: ΔEele: electrostatic energy in the gas phase; ΔEvdW: van der Waals energy; ΔEgas: total gas phase energy, which is the sum of ΔEele and ΔEvdW; ΔGpol: polar solvation free energy; ΔGnonpolar: nonpolar solvation free energy; ΔGsol: sum of nonpolar (ΔGnonpolar) and polar (ΔGpolar) contributions to salvation; ΔGbind: total binding free energy; cStandard deviation of mean values. 为了进一步了解每个残基在结合过程中的作用,我们用MM-PBSA方法对3个复合物体系中(AChE/M1~AChE/M3)每个残基的结合自由能进行了能量分解,如图 5(a)所示,标注出的是ΔGbind小于约-3.5 kJ·mol-1的残基,可以看出AChE与抑制剂相互作用的关键残基有:AChE/M1(Y70、V71、W84、W279、S286、I287、Y334)、AChE/M2(V71、W84、W279、F331、Y334)、AChE/M3(V71、Q74、W84、N85、W279、F290、F330、F331、Y334),这与实验上[20]以及其它理论研究[24]的结果相一致。表 2中比较了每一个能量项(ΔEele、ΔEvdW、ΔGpol、ΔGnonpolar)对总的自由能(ΔGbind)的贡献。通过分析表 2可以看出,范德华作用(ΔEvdW)对AChE与抑制剂的结合起主导作用(-241.17、-234.72、-200.58 kJ·mol-1)。极性溶剂化能(ΔGpol)对结合是不利的,尽管气相的静电能(ΔEele)对结合是有利的,但也不能完全抵消极性溶剂化自由能对结合的不利影响[50-51]。另外,非极性溶剂化能(ΔGnonpolar)也有较小的贡献。
 图5
AChE/抑制剂复合物体系中抑制剂-残基之间的相互作用能谱(a)和3个复合物体系的主要残基(Y70、V71、Q74、W84、N85、W279、S286、I287、F290、F330、F331、Y334)的能量比较(b)
Figure5.
Decomposition of total interaction energy on a per-residue basis for the three proteininhibitor complexes (a) and comparison of interaction energies of the major residues (Y70, V71, Q74, W84, N85, W279, S286, I287, F290, F330, F331, and Y334) for the three AChE/inhibitor complexes (b)
图5
AChE/抑制剂复合物体系中抑制剂-残基之间的相互作用能谱(a)和3个复合物体系的主要残基(Y70、V71、Q74、W84、N85、W279、S286、I287、F290、F330、F331、Y334)的能量比较(b)
Figure5.
Decomposition of total interaction energy on a per-residue basis for the three proteininhibitor complexes (a) and comparison of interaction energies of the major residues (Y70, V71, Q74, W84, N85, W279, S286, I287, F290, F330, F331, and Y334) for the three AChE/inhibitor complexes (b)
为了更好地验证3个抑制剂的生物活性,如图 5所示,我们深入分析了关键残基与抑制剂之间的相互作用。从图 5(a)中可以看出,对于AChE/M1、AChE/M2、AChE/M3复合物体系,这些关键残基包括Y70、V71、Q74、W84、N85、W279、S286、I287、F290、F330、F331和Y334。在这些残基中,主要残基W84、W279、Y334与抑制剂(M1、M2、M3)结合自由能的总和分别为-31.13、-30.17、-25.36 kJ·mol-1,使3个抑制剂的生物活性顺序为M1>M2>M3。图 5(b)表示关键残基与3个抑制剂的结合自由能图,残基Y334与抑制剂的结合自由能明显弱于残基W279与抑制剂间的结合自由能,后者更能有效地区分3个抑制剂的生物活性。
通过表 2得知,范德华作用对复合物的结合起主导作用,我们对范德华作用进行了残基自由能分解,结果如图 6(a)所示,列出的是ΔEvdW小于约-3.5 kJ·mol-1的残基。由表 2可以看出,抑制剂M1与AChE之间的范德华相互作用(ΔEvdW:-241.17 kJ·mol-1)明显强于抑制剂M2(或M3)与AChE之间的范德华相互作用(-234.72 (或-200.58) kJ·mol-1),使得3个抑制剂的生物活性顺序为M1>M2>M3。如图 6(a)所示,抑制剂M1与关键残基(Y70、V71、D72、S81、W84、Y121、W279、F330、F331、Y334)之间的范德华相互作用,尤其是残基Y70、W84、W279、Y334,对结合自由能有较大的贡献。从图 6(a)可以看出,尽管抑制剂M2和M3与残基Y334之间的范德华相互作用相对更强,但是抑制剂M1与残基W279之间的范德华相互作用远远强于M2(或M3)与W279之间的范德华相互作用。抑制剂M1、M2、M3与这些关键残基(Y70、V71、D72、Q74、S81、W84、Y121、W279、F330、F331、Y334)范德华相互作用之和分别为-84.10、-85.22、-70.12 kJ·mol-1,与3个抑制剂的生物活性大小顺序(M1(或M2)>M3)相一致。以上分析表明了抑制剂与残基之间的范德华相互作用,尤其是残基W279和Y334,对结合自由能以及区分3个抑制剂的生物活性上有很大的贡献。
为了揭示静电相互作用对抑制剂M1、M2、M3与AChE结合亲和力的影响,我们用LIGPLOT[52]程序分析了抑制剂与AChE之间的氢键作用和疏水相互作用。如图 7所示,结合态AChE周围的残基与结合自由能残基分解分析中得到的残基是一致的。从图 7(a)中可以看出,在AChE/M1体系中,存在一条氢键,形成于残基S286的氧原子(O)与M1上的氮原子(N9)之间。如表 3所示,这条氢键的占有率高达90.36%,从图 7(d)可以看出,这条氢键(O(S286)-N9(M1))在MD模拟过程中是趋于稳定的,平均距离是0.295 nm(表 3)。在AChE/M2体系中,如图 7(b)所示,在残基D72上的氮原子(N)与M2上的氮原子(N25)之间形成了一条占有率为31.56%的氢键,这条稳定的氢键(N(D72)-N25(M2))(图 7(d))的平均距离为0.334 nm(见表 3)。同样的,在AChE/M3体系中,如图 7(c)所示,残基D72上的氮原子(N)与M3上的氧原子(O26)之间形成了氢键,占有率为40.94%,这条氢键(N(D72)-O26(M3))稳定后的平均距离是0.323 nm(见表 3)。为了考察每个残基的静电相互作用对抑制剂与AChE结合的细节,我们对静电相互作用进行了残基自由能分解,结果如图 6(b)所示,列出的是ΔEele小于约-3.5 kJ·mol-1的残基(Y70、D72、E73、E199、D276、D285、S286)。从图 6(b)可以看出,虽然M3与残基D72之间的静电相互作用很强,但仍比M1与残基S286之间的静电相互作用要弱,这与上述的氢键分析结果是一致的。抑制剂M1、M2、M3分别与关键残基(D72和S286)的净静电相互作用(ΔEele+ΔGpol)之和分别为3.60、9.92和10.17 kJ·mol-1,虽然不利于抑制剂与AChE的结合,但与抑制剂的生物活性相一致。上述分析表明,M1与残基S286之间形成的氢键有利于抑制剂稳定在结合位点。
 图7
氢键和疏水作用二维图:虚线代表氢键, 穗状代表与3个抑制剂形成疏水作用的残基(a) AChE/M1; (b) AChE/M2;
(c) AChE/M3; (d)抑制剂与AChE之间形成的氢键的距离随MD模拟的时间变化, 其中, M1与残基S286的距离曲线上移了0.05 nm; M2与残基D72的距离曲线上移了0.2 nm
Figure7.
2D representation of hydrogen bonds and hydrophobic interactions: Dashed lines represent hydrogen bonds, and spiked residues form hydrophobic interactions for complexes AChE/M1 (a), AChE/M2 (b), and AChE/M3 (c); (d) Interatomic distances with the MD time evolution show the stability of hydrogen bonds between three inhibitors and AChE; M1: Curve of S286 is shifted upward by 0.05 nm; M2: Curve of D72 is shifted upward by 0.2 nm
图7
氢键和疏水作用二维图:虚线代表氢键, 穗状代表与3个抑制剂形成疏水作用的残基(a) AChE/M1; (b) AChE/M2;
(c) AChE/M3; (d)抑制剂与AChE之间形成的氢键的距离随MD模拟的时间变化, 其中, M1与残基S286的距离曲线上移了0.05 nm; M2与残基D72的距离曲线上移了0.2 nm
Figure7.
2D representation of hydrogen bonds and hydrophobic interactions: Dashed lines represent hydrogen bonds, and spiked residues form hydrophobic interactions for complexes AChE/M1 (a), AChE/M2 (b), and AChE/M3 (c); (d) Interatomic distances with the MD time evolution show the stability of hydrogen bonds between three inhibitors and AChE; M1: Curve of S286 is shifted upward by 0.05 nm; M2: Curve of D72 is shifted upward by 0.2 nm
 表 3
MD模拟过程中抑制剂与AChE之间形成的氢键
Table 3.
Hydrogen bonds formed between inhibitors and AChE during MD simulations
表 3
MD模拟过程中抑制剂与AChE之间形成的氢键
Table 3.
Hydrogen bonds formed between inhibitors and AChE during MD simulations
System Donor Acceptor Occupancy / % Distance / nm (SDa) Angle/(°)(SD) AChE/Ml :286@O :535@N9 90.36 0.295(0.017) 29.29(13.34) AChE/M2 :535@N25 :72@N 31.56 0.334(0.011) 19.92(10.28) AChE/M3 :535@O26 :72@N 40.94 0.323(0.017) 24.75(11.95) aStandard deviation of mean values. 基于上述分析,我们从表 2、图 6(a)、图 7中可以看出,在AChE/M1~M3体系中,抑制剂与AChE之间有较强的范德华相互作用,特别是AChE上的残基W279与抑制剂二氮杂咔唑母体、残基Y334与抑制剂上苯环的关键作用。抑制剂M1、M2、M3与这2个关键残基(W279和Y334)的范德华相互作用之和分别为-31.17、-34.60、-24.10 kJ·mol-1。另外,抑制剂M1与残基S286之间的静电相互作用要强于抑制剂M2/M3与残基D72之间的静电相互作用。通过比较AChE/M1、AChE/M2、AChE/M3复合物体系的结构和相互作用,可以看出母体二氮杂咔唑的重要性。如果二氮杂咔唑与含有一个苯环的适当的取代基相连,像抑制剂M1一样,就会在残基W279与抑制剂二氮杂咔唑母体之间产生较强的范德华相互作用,在残基S286与二氮杂咔唑母体上的唑氮之间产生较强的氢键作用,形成的二氮杂咔唑衍生物抑制剂对AChE就可能有较强的抑制活性。上述分析揭示了残基S286对M1与AChE的结合起有利作用,主要关键残基W279和Y334在抑制剂与AChE的结合中和区分3个抑制剂的生物活性中发挥了重要的作用。
3 结论
本文通过分子对接、分子动力学模拟和MM-PBSA能量计算方法,从原子水平上研究了3个1,7-二氮杂咔唑衍生物抑制剂与AChE的相互作用,并揭示了1,7-二氮杂咔唑衍生物抑制剂对AChE的抑制机理和3个抑制剂之间的生物活性差别。模拟结果表明复合物中稳定的氢键可以使抑制剂与AChE之间产生较强的静电作用,有利于两者的结合。结合能的计算结果与抑制剂的实验生物活性数据(IC50)吻合较好,证实了抑制剂的生物活性顺序为M1>M2>M3。结果表明,抑制剂与残基S286之间形成的氢键作用有助于抑制剂与AChE的结合。范德华相互作用对AChE/M1~M3体系的结合自由能有很大的贡献。主要关键残基W279和Y334对于抑制剂与AChE的结合及区分3个抑制剂的生物活性大小起了决定性作用。本文的工作有助于理解AChE抑制剂的抑制机理,并为设计新型的有潜力的AChE抑制剂提供了有价值的信息。
-
-
[1]
Mussele S V D, Bastard N L, Vermeiren Y, et al. Int. J. Geriatr. Psychiatr., 2013, 28(3):265-275 doi: 10.1002/gps.v28.3
-
[2]
Sperling R A, Dickerson B C, Pihlajamaki M, et al. Neuromol. Med., 2010, 12(1):27-43 doi: 10.1007/s12017-009-8109-7
-
[3]
Nestor P J, Fryer T D, Hodges J R. NeuroImage., 2006, 30 (3):1010-1020 doi: 10.1016/j.neuroimage.2005.10.008
-
[4]
Bartus R T, Dean R L, Beer B, et al. Science, 1982, 217 (4558):408-414 doi: 10.1126/science.7046051
-
[5]
Terry R D, Masliah E, Salmon D P, et al. Ann. Neurol., 1991, 30(4):572-580 doi: 10.1002/(ISSN)1531-8249
-
[6]
Francis P T, Palmer A M, Snape M, et al. J. Neurol. Neurosur. Ps., 1999, 67(4):558-558 doi: 10.1136/jnnp.67.4.558
-
[7]
Talesa V N. Mech. Ageing Dev., 2001, 122(16):1961-1969 doi: 10.1016/S0047-6374(01)00309-8
-
[8]
Alipour M, Khoobi M, Moradi A, et al. Eur. J. Med. Chem., 2014, 82:536-544 doi: 10.1016/j.ejmech.2014.05.056
-
[9]
Liu S, Shang R, Shi L, et al. Eur. J. Med. Chem., 2014, 81: 237-244 doi: 10.1016/j.ejmech.2014.05.020
-
[10]
Akrami H, Mirjalili B F, Khoobi M, et al. Eur. J. Med. Chem., 2014, 84:375-381 doi: 10.1016/j.ejmech.2014.01.017
-
[11]
Wang C, Wu Z, Cai H, et al. Bioorg. Med. Chem. Lett., 2015, 25(22):5212-5216 doi: 10.1016/j.bmcl.2015.09.063
-
[12]
Hong C, Luo W, Yao D, et al. Bioorg. Med. Chem., 2014, 22 (12):3213-3219 doi: 10.1016/j.bmc.2014.03.045
-
[13]
Giacobini E. Neurochem. Res., 2003, 28(3):515-522
-
[14]
Xie S S, Wang X B, Jiang N, et al. Eur. J. Med. Chem., 2015, 95:153-165 doi: 10.1016/j.ejmech.2015.03.040
-
[15]
Alpan A S, Parlar S, Carlino L, et al. Bioorgan. Med. Chem., 2013, 21(17):4928-4937 doi: 10.1016/j.bmc.2013.06.065
-
[16]
Mohammadi-Khanaposhtani M, Saeedi M, Zafarghandi N S, et al. Eur. J. Med. Chem., 2015, 92:799-806 doi: 10.1016/j.ejmech.2015.01.044
-
[17]
Asadipour A, Alipour M, Jafari M, et al. Eur. J. Med. Chem., 2013, 70:623-630 doi: 10.1016/j.ejmech.2013.10.024
-
[18]
Gao X, Zhou C, Liu H, et al. J. Enzyme Inhib. Med. Chem., 2017, 32(1):146-152 doi: 10.1080/14756366.2016.1243534
-
[19]
Liu H, Liu L, Gao X, et al. Eur. J. Med. Chem., 2017, 126: 810-822 doi: 10.1016/j.ejmech.2016.12.003
-
[20]
Gazzard L, Williams K, Chen H F, et al. J. Med. Chem., 2015, 58(12):5053-5074 doi: 10.1021/acs.jmedchem.5b00464
-
[21]
Niu C, XU Y, Xu Y, et al. J. Phys. Chem. B, 2005, 109(49): 23730-23738 doi: 10.1021/jp0552877
-
[22]
Puiatti M, Borioni J L, Vallejo M G, et al. J. Mol. Graph. Model., 2013, 44:136-144 doi: 10.1016/j.jmgm.2013.05.009
-
[23]
Zhu X L, Yu N X, Hao G F, et al. J. Mol. Graph. Model., 2013, 41:55-60 doi: 10.1016/j.jmgm.2013.01.004
-
[24]
Zhou A, Hu J, Wang L, et al. J. Mol. Model., 2015, 21(10): 277 doi: 10.1007/s00894-015-2797-8
-
[25]
Shi J, Tu W, Luo M, et al. Mol. Simul., 2016, 43(2):102-109
-
[26]
Mohammadi T, Ghayeb Y. J. Biomol. Struct. Dyn., 2017:1-13
-
[27]
Hossain T, Saha A, Mukherjee A. J. Biomol. Struct. Dyn., 2017:1-12
-
[28]
El-Malah A, Gedawy E M, Kassab A E, et al. Arch. Pharm. Chem. Life Sci., 2014, 347(2):96-103 doi: 10.1002/ardp.v347.2
-
[29]
Molinuevo J L, Berthier M L, Rami L. Arch. Gerontol. Geriatr., 2011, 52(1):18-22 doi: 10.1016/j.archger.2009.11.004
-
[30]
Knowles J. Core Evidence, 2006, 1(3):195-219
-
[31]
Marco L, Carreiras M C. Recent Pat. CNS Drug. Discovery, 2006, 1(1):105-111 doi: 10.2174/157488906775245246
-
[32]
Wolfson C, Oremus M, Shukla V, et al. Clin. Ther., 2002, 24 (6):862-886 doi: 10.1016/S0149-2918(02)80004-2
-
[33]
Bono G F, Simo-Silva D P, Batistela M S, et al. Neurochem. Int., 2015, 81:57-62 doi: 10.1016/j.neuint.2014.12.009
-
[34]
Kryger G, Silman I, Sussman J L. Structure, 1999, 7(3):297-307 doi: 10.1016/S0969-2126(99)80040-9
-
[35]
Larkin M, Blackshields G, Brown N P, et al. Bioinformatics, 2007, 23:2947-2948 doi: 10.1093/bioinformatics/btm404
-
[36]
Morris G M, Goodsell D S, Halliday R S, et al. J. Comput. Chem., 1998, 19(14):1639-1662 doi: 10.1002/(ISSN)1096-987X
-
[37]
Cornell W D, Cieplak P, Bayly C I, et al. J. Am. Chem. Soc., 1995, 117(19):5179-5197 doi: 10.1021/ja00124a002
-
[38]
Wang J, Wolf R M, Caldwell J W, et al. J. Comput. Chem., 2004, 25(9):1157-1174 doi: 10.1002/(ISSN)1096-987X
-
[39]
Bayly C I, Cieplak P, Cornell W, et al. J. Phys. Chem., 1993, 97(40):10269-10280 doi: 10.1021/j100142a004
-
[40]
Jorgensen W L, Chandrasekhar J, Madura J D, et al. J. Chem. Phys., 1983, 79(2):926-935 doi: 10.1063/1.445869
-
[41]
Rycaert J P, Ciccotti G, Berendsen H J C. J. Comput. Phys., 1977, 23:327-341 doi: 10.1016/0021-9991(77)90098-5
-
[42]
York D M, Darden T A, Pedersen L G. J. Chern. Phys., 1993, 99(10):8345-8348 doi: 10.1063/1.465608
-
[43]
Kumari R, Kumar R, Open Source Drug Discovery Consortium, et al. J. Chem. Inf. Model., 2014, 54(7):1951-1962 doi: 10.1021/ci500020m
-
[44]
Saíz-Urra L, Cabrera M A, Froeyen M. J. Mol. Graph. Model., 2011, 29(5):726-739 doi: 10.1016/j.jmgm.2010.12.005
-
[45]
El-Barghouthi M I, Jaime C, Al-Sakhen N A, et al. J. Mol. Struct. THEOCHEM, 2008, 853(1):45-52
-
[46]
Fajer P, Fajer M, Zawrotny M, et al. Methods Enzymol., 2015, 563:623-642 doi: 10.1016/bs.mie.2015.07.030
-
[47]
Sa R, Fang L, Huang M, et al. J. Phys. Chem. A, 2014, 118: 9113-9119 doi: 10.1021/jp5064319
-
[48]
Jamshidi S, Rafii-Tabar H, Jalili S. Mol. Simul., 2013, 40: 469-476
-
[49]
Lobanov M Y, Bogatyreva N S, Galzitskaya O V. Mol. Biol., 2008, 42(4):623-628 doi: 10.1134/S0026893308040195
-
[50]
Hu G D, Zhu T, Zhang S L, et al. Eur. J. Med. Chem., 2010, 45(1):227-235 doi: 10.1016/j.ejmech.2009.09.048
-
[51]
Wu E L, Han K L, Zhang J Z H. Chem. Eur. J., 2008, 14 (28):8704-8714 doi: 10.1002/chem.v14:28
-
[52]
Laskowski R A, Swindells M B. J. Chem. Inf. Model., 2011, 51:2778-2786 doi: 10.1021/ci200227u
-
[1]
-
图 5 AChE/抑制剂复合物体系中抑制剂-残基之间的相互作用能谱(a)和3个复合物体系的主要残基(Y70、V71、Q74、W84、N85、W279、S286、I287、F290、F330、F331、Y334)的能量比较(b)
Figure 5 Decomposition of total interaction energy on a per-residue basis for the three proteininhibitor complexes (a) and comparison of interaction energies of the major residues (Y70, V71, Q74, W84, N85, W279, S286, I287, F290, F330, F331, and Y334) for the three AChE/inhibitor complexes (b)
In (a), the curves of AChE/M2 and AChE/M3 are shifted right by 400 and 800, respectively
图 6 3个抑制剂与AChE复合物抑制剂-残基的范德华作用(a)和3个抑制剂与AChE复合物抑制剂-残基的静电相互作用(b)
Figure 6 Van der Waals interaction energy spectra of inhibitor-residue pair in AChE/inhibitor complexes (a) and electrostatic interaction energy spectra of inhibitor-residue pair in AChE/inhibitor complexes (b)
In (a) and (b), the curves of AChE/M2 and AChE/M3 are shifted right by 400 and 800, respectively
图 7 氢键和疏水作用二维图:虚线代表氢键, 穗状代表与3个抑制剂形成疏水作用的残基(a) AChE/M1; (b) AChE/M2; (c) AChE/M3; (d)抑制剂与AChE之间形成的氢键的距离随MD模拟的时间变化, 其中, M1与残基S286的距离曲线上移了0.05 nm; M2与残基D72的距离曲线上移了0.2 nm
Figure 7 2D representation of hydrogen bonds and hydrophobic interactions: Dashed lines represent hydrogen bonds, and spiked residues form hydrophobic interactions for complexes AChE/M1 (a), AChE/M2 (b), and AChE/M3 (c); (d) Interatomic distances with the MD time evolution show the stability of hydrogen bonds between three inhibitors and AChE; M1: Curve of S286 is shifted upward by 0.05 nm; M2: Curve of D72 is shifted upward by 0.2 nm
表 1 3个1, 7-二氮杂咔唑衍生物抑制剂的结构和IC50值
Table 1. Structures and bioactivity of three 1, 7-diazacarbazole derivative inhibitors to AChE

Inhibitor R IC50 / (nmol·mL-1) M1 
0.890 M2 
4.77 M3 
13.3 表 2 通过MM-PBSA方法计算得到的复合物的结合自由能
Table 2. Binding free energy components for the proteininhibitor complexes by using the MM-PBSA methoda
Componentb AChE/M1(SDc) AChE/M2(SD) AChE/M3(SD) ΔEele -56.48(15.65) -22.51(13.22) -27.41(19.41) ΔEvdw -241.17(9.33) -234.72(9.46) -200.58(10.04) ΔEgas -297.65(16.57) -257.23(13.93) -227.99(25.40) ΔEpol 181.42(13.77) 155.14(19.71) 152.47(28.62) ΔGnonpolar -28.16(0.63) -27.32(0.59) -26.99(0.88) ΔGsol 153.26(13.64) 127.86(19.87) 125.52(28.53) ΔGbind -144.35(15.02) -129.37(19.08) -102.47(19.00) IC50 0.890 4.77 13.3 aAll values are given in kJ·mol-1; bComponents: ΔEele: electrostatic energy in the gas phase; ΔEvdW: van der Waals energy; ΔEgas: total gas phase energy, which is the sum of ΔEele and ΔEvdW; ΔGpol: polar solvation free energy; ΔGnonpolar: nonpolar solvation free energy; ΔGsol: sum of nonpolar (ΔGnonpolar) and polar (ΔGpolar) contributions to salvation; ΔGbind: total binding free energy; cStandard deviation of mean values. 表 3 MD模拟过程中抑制剂与AChE之间形成的氢键
Table 3. Hydrogen bonds formed between inhibitors and AChE during MD simulations
System Donor Acceptor Occupancy / % Distance / nm (SDa) Angle/(°)(SD) AChE/Ml :286@O :535@N9 90.36 0.295(0.017) 29.29(13.34) AChE/M2 :535@N25 :72@N 31.56 0.334(0.011) 19.92(10.28) AChE/M3 :535@O26 :72@N 40.94 0.323(0.017) 24.75(11.95) aStandard deviation of mean values. -

 扫一扫看文章
扫一扫看文章
计量
- PDF下载量: 18
- 文章访问数: 2001
- HTML全文浏览量: 478







 下载:
下载:






 下载:
下载:

