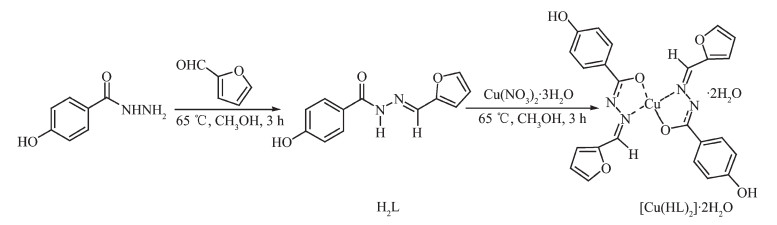
 图 Scheme 1
图 Scheme 1

Citation: LIU Xiang-Rong, SUN Xiu-Chao, YANG Zai-Wen, ZHAO Shun-Sheng, YANG Shui-Lan, YAN Sen. 2-Furancarbaldehyde-4-hydroxy-benzoylhydrazone and Its Cu (Ⅱ) Complex: Crystal Structures and Binding Ability with CT-DNA[J]. Chinese Journal of Inorganic Chemistry, 2016, 32(2): 250-258. doi: 10.11862/CJIC.2016.037

2-呋喃甲醛-4-羟基苯甲酰腙及其Cu (Ⅱ) 配合物的晶体结构及与CT-DNA的结合性能
-
关键词:
- 酰腙化合物
- / Cu (Ⅱ) 配合物
- / 晶体结构
- / CT-DNA
- / 微量热
English
2-Furancarbaldehyde-4-hydroxy-benzoylhydrazone and Its Cu (Ⅱ) Complex: Crystal Structures and Binding Ability with CT-DNA
-
Key words:
- hydrazone compounds
- / Cu (Ⅱ) complex
- / crystal structure
- / CT-DNA
- / microcalorimetry
-
0 引言
酰腙化合物分子结构中含有生物活性基团-CONHN=CH-,不仅具有优良的抑菌、杀菌、抗肿瘤、除草性等生物药理活性,而且分子中的O、N均可作为配位原子,具有较强的配位能力和多样的配位方式[1-2],尤其是酰腙的过渡金属配合物,由于融合了有机和无机单元,在生物医学、发光材料和催化领域应用比较广泛[3-5]。据文献报道[6-8],大部分具有生物活性的化合物易以插入方式与病毒或肿瘤的DNA作用,从而达到抗病毒、抗肿瘤的目的。因此,本文拟将活性基团呋喃引入酰腙分子中实现活性叠加,获得更具抑、杀菌能力的新物质,故以2-呋喃甲醛和4-羟基苯甲酰肼为原料缩合得到2-呋喃甲醛-4-羟基苯甲酰腙,并以其为配体制备Cu (Ⅱ) 配合物。通过X射线单晶衍射和元素分析确定配体及配合物的晶体结构,利用热重实验分析它们的热分解过程,采用紫外吸收光谱探索配体及配合物与CT-DNA的作用模式,并借助微量热法测定其与CT-DNA相互作用过程的热效应,以此评价配体和配合物与CT-DNA的结合能力。
1 实验部分
1.1 主要试剂与仪器
2-呋喃甲醛、4-羟基苯甲酰肼、甲醇均为分析纯, CT-DNA (小牛胸腺DNA) 购自于美国Sigma公司。
XT4-100B熔点仪;PE-2400-Ⅱ元素分析仪;BRUKER SMART APEX Ⅱ CCD单晶衍射仪;METTLER-TOLEDO TG-DSC1 HT热重分析仪 (N2流量为100.00 mL·min-1);TU-1900型紫外-可见分光光度计;SETARAM C80微量热仪。
1.2 配体H2L的合成及单晶培养
配体和配合物的合成路线见Scheme 1。
称取0.153 2 g (1 mmol) 4-羟基苯甲酰肼溶于15.0 mL甲醇中,量取90 μL (1 mmol) 2-呋喃甲醛,慢慢滴加到4-羟基苯甲酰肼中,65 ℃搅拌下水浴回流3 h,冷却至室温后过滤,静置约1周后产生棕黄色块状晶体 (可供测试用),产率67.45%。m.p. 248~249 ℃。元素分析按C12H10N2O3计算的理论值 (%):C 62.60,H 4.38,N 12.17;实验值:C 62.61,H 4.43,N 11.81。
1.3 配合物1的合成及单晶培养
称取0.034 7 g (0.15 mmol) 配体H2L溶于10.0 mL甲醇中,待其完全溶解后,慢慢向其滴加0.036 3 g (0.15 mmol) Cu (NO3)2·3H2O的甲醇溶液,65 ℃搅拌下水浴回流3h,冷却至室温后过滤,静置一段时间后产生墨绿色块状晶体 (可供测试用)。m.p.>300 ℃。元素分析按CuC24H22N4O8计算的理论值 (%):C 51.61,H 3.94,N 10.04;实验值:C 51.22,H 3.87,N 9.75。
1.4 晶体结构测试
选取尺寸分别为0.37 mm×0.29 mm×0.13 mm的H2L的单晶和0.35 mm×0.21 mm×0.12 mm的1的单晶置于Bruker APEX-ⅡCCD单晶衍射仪上,在296(2) K下用经石墨单色器单色化的Mo Kα射线 (λ=0.071 073 nm),以φ-ω扫描方式,分别在2.68°~25.10°(H2L) 和2.37°~25.10°(1) 范围内收集单晶衍射数据,然后用SAINT程序进行还原。衍射强度数据经SADABS程序作经验吸收校正后用SHELXS程序[9]由直接法解出晶体结构,对非氢原子坐标及其各向异性热参数用SHELXL程序[10]进行全矩阵最小二乘法修正,与其相连的氢原子由理论加氢法得到。晶体学数据详见表 1。
Compound H2L 1 Chemical formula C12H10N2O3 CuC24H18N4O6-2H20 Formula weight 230.22 558.00 Crystal system Orthorhombic Monoclinic Space group Pna21 P21/c a/nm 0.958 3(2) 1.128 1(6) b/nm 1.117 5(3) 0.611 4(3) c/nm 1.034 5(3) 1.800 6(10) β/(°) 107.265(11) V/nm3 1.107 8(5) 1.186 0(11) Z 4 2 Dc/(g·cm-3) 1.380 1.563 F(000) 480 574 Reflections collected, unique 5 185, 1 958 (Rint=0.028 4) 5 691, 2 113 (Rint=0.149 2) Largest cliff, peak and hole/(e·nm-3) 129 and-198 396 and-437 Goodness of fit on F2 1.082 1.112 Final R indices [I>2σ(I)] R1=0.038 0, wR2=0.092 0 R1=0.095 3, wR2=0.123 7 R indices (all data) R1=0.045 7, wR2=0.097 2 R1=0.171 8, wR2=00149 1 CCDC:1030242,H2L;1401955,1。
1.5 热重实验
准确称取 (10.0±0.05) mg H2L或配合物1放入氧化铝坩埚内,升温程序设置为室温至800 ℃,升温速率为5.00、10.00和15.00 ℃·min-1,在氮气气氛下进行热重分析。
1.6 紫外吸收光谱
将3 mL 0.01 mol·L-1的Tris-HCl缓冲溶液 (pH=7.90) 加入参比比色皿中,向样品比色皿中加入等体积现配制的1×10-4 mol·L-1配体溶液,用微量进样器分别往参比比色皿和样品比色皿中加入相同体积 (50 μL) 100 mg·L-1的CT-DNA溶液,使配体与CT-DNA的浓度比值逐渐减小,连续加入5次,每次间隔约需5 min混合均匀,扫描波长范围为250~500 nm;配合物采用同样的测试方法。
1.7 微量热实验
准确称取2.0 mg的配体放入样品池下层,分别向参比池和样品池下层各加0.01 mol·L-1的Tris-HCl缓冲溶液 (pH=7.90) 2 mL,再分别向参比池和样品池的上层各加1 mL 100 mg·L-1的CT-DNA溶液,25 ℃下,用微量热仪测量配体与CT-DNA作用的热量变化;用同样的方法测量配合物与CT-DNA作用过程的热量变化。
2 结果与讨论
2.1 晶体结构
从表 1可以看出,H2L的晶体属于正交晶系,空间群为Pna21,其分子结构见图 1,主要键长及键角见表 2。从表 2可以看出,所列出的键长和键角的数据都在正常范围之内,H2L中苯环与呋喃环的二面角为32.5°,表明两个环不在同一平面上。N2-C8键长为0.128 2(3) nm,小于N1-C7(0.135 0(3) nm) 的键长,说明N2和C8之间形成了双键[11]。分子中O2-C7键长为0.123 6(3) nm,属于典型的C=O双键,证明配体是以酮羰基形式存在的。图 2为配体的氢键图,从图 2可以看出配体分子间通过氢键N-H…O (N1-H1…O2i氢键:H1…O2i 0.213 nm,N1…O2i 0.291 4(2) nm,N1-H1…O2i 151.1°) 和氢键O-H…N (O1-H1A…N2ii氢键:H1A…N2ii 0.216 nm,O1···N2ii 0.296 9(3) nm,O1-H1A…N2ii 169.5°) 的弱作用力拓展为如图 3所示波浪形堆积结构。氢键键长和键角见表 3。
H2L 02-C7 0.123 6(3) N1-C7 0.135 0(3) N2-C8 0.128 2(3) N1-N2 0.138 2(2) C6-C7 0.147 5(3) C8-C9 0.142 3(3) C7-N1-N2 118.19(17) 02-C7-N1 120.36(19) C8-N2-N1 115.41(18) N1-C7-C6 118.17(18) 03-C9-C8 119.2(2) C10-C9-C8 129.9(2) 1 02-C7 0.129 9(9) N1-C7 0.133 2(9) N2-C8 0.128 4(9) N1-N2 0.141 8(7) C6-C7 0.147 5(10) C8-C9 0.143 5(10) Cul-02 0.191 2(5) Cul-N2 0.192 5(6) Nl-N2-Cul 115.1(4) C7-N1-N2 107.4(6) 02-C7-N1 124.4(7) C7-02-Cul 111.4(5) 02-Cul-N2 81.6(2) O2-Cul-O2i 180.0 N2-Cul-N2i 180.0 Symmetry code: i1-x, -y, -z D-H…A d(D-H) d(H…A) d(D…A) ∠DHA N1-H1…O2i 0.086 0.213 0.291 4(2) 151.1 O1-H1A…N2ii 0.082 0.216 0.296 9(3) 169.5 Symmetry codes: ix-0.5, 0.5-y, z; ii2-x, -y, z-0.5 配合物1属于单斜晶系,P21/c空间群,其分子结构见图 4,主要键长及键角见表 2,从表 2可以看出所列出的键长和键角的数据都在正常范围之内。1的晶体单胞结构中包含1个Cu2+、2个配体负离子,并含有结晶水分子。1属于双齿配位,每个配体负离子上的亚胺N和羰基O都参与配位,形成四配位的单核结构,Cu2+所在环的内角求和是539.9°,说明Cu2+与配位原子组成了闭合的五元环。O2-Cu1-O2i和N2-Cu1-N2i键角均为180.0°,表明Cu2+周围的2个五元环几乎处于同一平面。配位后分子中的O2-C7键长变长,N1-C7键长变短,且配合物中N1-C7键长为0.133 2(9) nm,比吡啶环中N-C双键 (0.135 2 nm) 短,说明配体是以烯醇形式配位的。围绕Cu2+的Cu1-O2键长为0.191 2(5) nm,Cu1-N2键长为0.192 5(6) nm,与类似结构的铜配合物的键长基本一致[12-13]。
如图 5所示,配合物1中苯环上羟基的O1、H1A和结晶水中的O4形成氢键O1-H1A…O4ii(0.272 4(9) nm,151.9°),结晶水中的O4、H4B、H4C和N1、O1形成氢键O4-H4C…N1iii(0.301 3(10) nm,150(7)°) 和O4-H4B…O1iv(0.287 1(10) nm,157(7)°),单体分子通过以上3种弱氢键作用相互连接成如图 6所示规则交叉排列的三维超分子结构。氢键参数详见表 4。
D-H…A d(D-H) d(H…A) d(D…A) ∠DHA O1-H1A…O4ii 0.082 0.197 0.272 4(9) 151.9 O4-H4C-N1iii 0.084 6(10) 0.225(4) 0.301 3(10) 150(7) O4-H4B…O1iv 0.084 9(10) 0.207(3) 0.287 1(10) 157(7) Symmetry codes: iix+1, y, z; iii1-x, 0.5+y, 0.5-z; iv1-x, 1-y, 1-z 2.2 热重分析
2.3 CT-DNA结合性能
2.2.2 配合物1热重分析
配合物1的热重分析曲线如图 9~10所示,配合物在3种升温速率下的DTG曲线很相似。从图 9可见配合物的热分解过程主要分为2个阶段:第1阶段分解的实验失重率为7.97%,该阶段是配合物中2分子结晶水的失去,第2阶段对应的实验失重率为60.16%,最大失重率对应的热分解温度配合物与配体接近,均大于290 ℃,说明配合物和配体的热稳定性都较高。利用公式 (1) 和 (2) 计算的配合物热分解过程的表观活化能也列于表 5。从表 5可以看出,配体和配合物的主要热分解阶段的表观活化能分别为:145.8 kJ·moL-1和264.1 kJ·moL-1。
2.2.1 配体H2L热重分析
配体H2L在5.00 ℃·min-1升温速率下的TG-DTG曲线如图 7所示,在升温速率分别为5.00、10.00和15.00 ℃·min-1时的DTG曲线见图 8。可以看出配体在3种升温速率下的热分解过程相似,都只有一个阶段,分解峰分别出现在291.50、303.55和310.89 ℃,表明配体在291.50 ℃以下可以稳定存在。升温速率为5.00 ℃·min-1时实验失重率为70.67%,计算值70.90%(与OH-C6H4-CO-NH-N=C-的百分含量相同),因此可推测热分解过程中C8-C9键断裂。
采用Kissinger和Ozawa公式[14]计算配体热分解过程表观活化能,计算公式分别见 (1) 和 (2) 式。
其中,Tp为分解峰的温度,A为指前因子,Ea为表观活化能,R为气体摩尔常数,β为升温速率,G(α) 为积分机理函数。利用公式 (1) 和 (2) 可求得表观活化能Ea及指前因子A,计算结果列于表 5。
Compound β/(℃·min-1) Tp/℃ Kissinger Eq. Ozawa Eq. Ea/(kJ·mol-1) lgA r Ea/(kJ·mol-1) r H2L 5.00 291.50 10.00 303.55 145.8 11.15 -1.000 0 147.8 -1.000 0 15.00 310.89 1 5.00 290.69 10.00 297.84 264.1 22.39 -0.998 9 260.2 -0.999 0 15.00 301.41 2.3.2 H2L和1与CT-DNA作用的微量热分析
图 15和图 16分别是配体及配合物与CT-DNA相互作用的微量热实验热谱图。从图 15可以看出配体与CT-DNA混合后迅速放热,在11.20 min时出现一个最大放热峰,35.10 min时结束,通过计算峰面积得出配体与CT-DNA作用的焓变值为-4.56 kJ·moL-1。从图 16可以看出配合物与CT-DNA混合后发生了与配体类似的放热过程,最大放热峰出现在12.36 min,在49.37 min时结束,配合物与CT-DNA作用的焓变值为-22.53 kJ·moL-1,从焓变值可知配合物与CT-DNA有更强的作用。由于配体和配合物与CT-DNA作用焓变的绝对值都小于30 kJ·moL-1,因此可认为配体和配合物与CT-DNA没有发生化学反应,不存在化学键[25-26],这也印证了2.3.1节中的结果,即配体和配合物与CT-DNA均以插入方式相互作用。
2.3.1 H2L和1与CT-DNA作用的紫外吸收光谱分析
配体和配合物与CT-DNA相互作用的紫外吸收光谱图分别见图 11和图 12,从图 11可以看出,随CT-DNA浓度的增加,配体体系的吸光度值逐渐减小 (0.42→0.38),在326 nm处发生了减色效应和微弱的红移现象,可认为配体与CT-DNA发生插入作用,这是由于CT-DNA碱基对π电子轨道与配体中π*电子空轨道发生耦合,从而导致π-π*跃迁能减小[15-19],产生红移现象,此外,配体插入CT-DNA后,长链DNA的“屏蔽”使π平面接受的光通量减少,也会导致减色效应[20-21]。图 12中配合物体系的吸光度也随CT-DNA浓度的增加而逐渐减小 (0.22→0.19,0.15→0.13),分别在297 nm和355 nm处发生了减色效应和微弱的红移现象,表明配合物也是以插入作用与CT-DNA结合,配合物与CT-DNA作用时,具有芳香性的配体会部分嵌入到DNA的相邻碱基对之间,这一插入作用是药物分子与DNA结合的一个重要模式,而且金属插入剂是这种结合模式极有用的探针[22]。
配体和配合物与CT-DNA的结合常数Kb可以通过收集在指定波长下的滴定曲线的吸光度并利用方程 (3)[23]求得,其中εa,εf和εb分别表示任意CT-DNA浓度下Tris-HCl缓冲溶液的摩尔消光系数、配体或配合物的摩尔消光系数和配体或配合物与CT-DNA完全键合后的摩尔消光系数[24]。以CDNA/(εa-εf) 对CCT-DNA作图,配体和配合物的CDNA/(εa-εf)-CDNA关系图分别见图 13和图 14,斜率与截距的比值即为样品与CT-DNA的结合常数Kb。通过计算,配体和配合物的结合常数分别为:1.98×106 L·moL-1和3.68×107 L·moL-1,表明配体和配合物与CT-DNA都有较强的插入作用[23],并且配合物与CT-DNA的结合能力大于配体。
3 结论
以2-呋喃甲醛和4-羟基苯甲酰肼为原料制备了配体2-呋喃甲醛-4-羟基苯甲酰腙 (H2L),并进一步制得其Cu (Ⅱ) 配合物[Cu (HL)2]·2H2O (1),利用元素分析和X射线单晶衍射确定了其结构。热重实验结果说明H2L和1的热稳定性都较高。通过紫外吸收光谱法和微量热法研究了H2L和1与CT-DNA的相互作用模式和强弱,结果表明H2L和1与CT-DNA皆以插入模式结合,作用过程放热,1与CT-DNA的结合能力强于H2L。
-
-
[1]
杨锐, 何水样, 武望婷, 等.化学学报, 2004, 62(20):2040-2044 doi: 10.3321/j.issn:0567-7351.2004.20.014YANG Rui, HE Shui-Yang, WU Wang-Ting, et al. Acta Chim. Sinica, 2004, 62(20):2040-2044 doi: 10.3321/j.issn:0567-7351.2004.20.014
-
[2]
Monfort M, Resino J, Ribas J, et al. Inorg. Chem., 2000, 3(9):2572-2576 http://www.ncbi.nlm.nih.gov/pubmed/11197011
-
[3]
任海仙, 唐静, 魏太保, 等.无机化学学报, 2007, 23(11):1907-1911 doi: 10.3321/j.issn:1001-4861.2007.11.011REN Hai-Xian, TANG Jing, WEI Tai-Bao, et al. Chinese J. Inorg. Chem., 2007, 23(11):1907-1911 doi: 10.3321/j.issn:1001-4861.2007.11.011
-
[4]
Schuetz S A, Silvernail C M, Incarvito C D. Inorg. Chem., 2004, 4(3):6203-6207
-
[5]
Adsule S, Barve V, Ahmed F, et al. J. Med. Chem., 2006, 49(24):7242-7246 doi: 10.1021/jm060712l
-
[6]
Liu S, Liu F, Yu X, et al. Bioorg. Med. Chem., 2006, 14:1425-1430 doi: 10.1016/j.bmc.2005.09.064
-
[7]
李玲, 董同义, 李修录, 等.药学学报, 1994, 29:128-131LI Ling, DONG Tong-Yi, LI Xiu-Lu, et al. Acta Pharm. Sin., 1994, 29:128-131
-
[8]
Cao S L, Feng Y P, Jiang Y Y, et al. Bioorg. Med. Chem.Lett., 2005, 15:1915-1917 doi: 10.1016/j.bmcl.2005.01.083
-
[9]
Sheldrick G M. SHELXS-97, Program for the Solution ofCrystal Structures, University of Göttingen, Germany, 1997.
-
[10]
Sheldrick G M. SHELXL-97, Program for the Refinement ofCrystal Structures, University of Göttingen, Germany, 1997.
-
[11]
龙德清, 陈胜胜, 陈辉.合成化学, 2006, 14(1):69-71 http://www.cqvip.com/QK/71135X/201107/21077207.htmlLONG De-Qing, CHEN Sheng-Sheng, CHEN Hui. Chinese J. Synth. Chem., 2006, 14(1):69-71 http://www.cqvip.com/QK/71135X/201107/21077207.html
-
[12]
吴琼洁, 陈小华, 蔡碧琼, 等.无机化学学报, 2012, 28(1):201-206 http://www.wjhxxb.cn/wjhxxbcn/ch/reader/view_abstract.aspx?flag=1&file_no=20120135&journal_id=wjhxxbcnWU Qiong-Jie, CHEN Xiao-Hua, CAI Bi-Qiong, et al. Chinese J. Inorg. Chem., 2012, 28(1):201-206 http://www.wjhxxb.cn/wjhxxbcn/ch/reader/view_abstract.aspx?flag=1&file_no=20120135&journal_id=wjhxxbcn
-
[13]
郑长征, 王燕燕, 王亮.无机化学学报, 2014, 30(7):1474-1480 http://www.wjhxxb.cn/wjhxxbcn/ch/reader/view_abstract.aspx?flag=1&file_no=20140637&journal_id=wjhxxbcnZHENG Chang-Zheng, WANG Yan-Yan, WANG Liang. Chinese J. Inorg. Chem., 2014, 30(7):1474-1480 http://www.wjhxxb.cn/wjhxxbcn/ch/reader/view_abstract.aspx?flag=1&file_no=20140637&journal_id=wjhxxbcn
-
[14]
HU Rong-Zu, GAO Sheng-Li, ZHAO Feng-Qi, et al. Thermal Analysis Kinetics. 2nd. Beijing: Science Press, 2008:79-120
-
[15]
高婷, 张万举, 王芳, 等.广州化工, 2010, 38(5):23-25GAO Ting, ZHANG Wan-Ju, WANG Fang, et al. Guangzhou Chem. Ind., 2010, 38(5):23-25
-
[16]
Wolfe A, Shimer G H, Meehan T. Biochem., 1987, 26:6392-6396 doi: 10.1021/bi00394a013
-
[17]
Pyle A M, Rehmann J P, Meshoyrer R, et al. J. Am. Chem.Soc., 1989, 111:3051-3058
-
[18]
Raman N, Sobha S. Inorg. Chem. Commun., 2012, 17:120-123 doi: 10.1016/j.inoche.2011.12.029
-
[19]
Eberhard J, Stoll I, Brockhinke R, et al. CrystEngComm, 2013, 15:4225-4248 doi: 10.1039/c2ce26388b
-
[20]
Rajput C, Rutkaite R, Swanson L, et al. Chem. Eur. J., 2006, 12:4611-4619 doi: 10.1002/(ISSN)1521-3765
-
[21]
Mabeswari P U, Palaniandavar M. J. Inorg. Biochem., 2004, 98:219-230 doi: 10.1016/j.jinorgbio.2003.09.003
-
[22]
Carter M T, Rodriguez M, Bard A J. J. Inorg. Biochem., 2008, 102:330-341 doi: 10.1016/j.jinorgbio.2007.09.006
-
[23]
朱莉, 彭斌, 凌友, 等.化学学报, 2008, 66(24):2705-2711 doi: 10.3321/j.issn:0567-7351.2008.24.008ZHU Li, PENG Bin, LING You, et al.Acta Chim. Sinica, 2008, 66(24):2705-2711 doi: 10.3321/j.issn:0567-7351.2008.24.008
-
[24]
Shi S, Geng X T, Zhao J, et al. Biochimie, 2010, 92:370-377 doi: 10.1016/j.biochi.2010.01.003
-
[25]
Solimani R. Biochim. Biophys. Acta, 1997, 1336(2):281-294 doi: 10.1016/S0304-4165(97)00038-X
-
[26]
Peter J, Ingermar W. J. Biochem. Biopphys. Methods, 1997, 35(103):103-114
-
[1]
-
表 1 H2L和1的晶体学数据
Table 1. Crystallographic data for H2L and 1
Compound H2L 1 Chemical formula C12H10N2O3 CuC24H18N4O6-2H20 Formula weight 230.22 558.00 Crystal system Orthorhombic Monoclinic Space group Pna21 P21/c a/nm 0.958 3(2) 1.128 1(6) b/nm 1.117 5(3) 0.611 4(3) c/nm 1.034 5(3) 1.800 6(10) β/(°) 107.265(11) V/nm3 1.107 8(5) 1.186 0(11) Z 4 2 Dc/(g·cm-3) 1.380 1.563 F(000) 480 574 Reflections collected, unique 5 185, 1 958 (Rint=0.028 4) 5 691, 2 113 (Rint=0.149 2) Largest cliff, peak and hole/(e·nm-3) 129 and-198 396 and-437 Goodness of fit on F2 1.082 1.112 Final R indices [I>2σ(I)] R1=0.038 0, wR2=0.092 0 R1=0.095 3, wR2=0.123 7 R indices (all data) R1=0.045 7, wR2=0.097 2 R1=0.171 8, wR2=00149 1 表 2 H2L和1的主要键长 (nm) 及键角 (°)
Table 2. Selected bond lengths (nm) and angles (°) for H2L and 1
H2L 02-C7 0.123 6(3) N1-C7 0.135 0(3) N2-C8 0.128 2(3) N1-N2 0.138 2(2) C6-C7 0.147 5(3) C8-C9 0.142 3(3) C7-N1-N2 118.19(17) 02-C7-N1 120.36(19) C8-N2-N1 115.41(18) N1-C7-C6 118.17(18) 03-C9-C8 119.2(2) C10-C9-C8 129.9(2) 1 02-C7 0.129 9(9) N1-C7 0.133 2(9) N2-C8 0.128 4(9) N1-N2 0.141 8(7) C6-C7 0.147 5(10) C8-C9 0.143 5(10) Cul-02 0.191 2(5) Cul-N2 0.192 5(6) Nl-N2-Cul 115.1(4) C7-N1-N2 107.4(6) 02-C7-N1 124.4(7) C7-02-Cul 111.4(5) 02-Cul-N2 81.6(2) O2-Cul-O2i 180.0 N2-Cul-N2i 180.0 Symmetry code: i1-x, -y, -z 表 3 H2L的氢键键长 (nm) 及键角 (°)
Table 3. Hydrogen bond distances (nm) and angles (°) of H2L
D-H…A d(D-H) d(H…A) d(D…A) ∠DHA N1-H1…O2i 0.086 0.213 0.291 4(2) 151.1 O1-H1A…N2ii 0.082 0.216 0.296 9(3) 169.5 Symmetry codes: ix-0.5, 0.5-y, z; ii2-x, -y, z-0.5 表 4 配合物1的氢键键长 (nm) 及键角 (°)
Table 4. Hydrogen bond distances (nm) and angles (°) of complex 1
D-H…A d(D-H) d(H…A) d(D…A) ∠DHA O1-H1A…O4ii 0.082 0.197 0.272 4(9) 151.9 O4-H4C-N1iii 0.084 6(10) 0.225(4) 0.301 3(10) 150(7) O4-H4B…O1iv 0.084 9(10) 0.207(3) 0.287 1(10) 157(7) Symmetry codes: iix+1, y, z; iii1-x, 0.5+y, 0.5-z; iv1-x, 1-y, 1-z 表 5 H2L和1在3种升温速率下的热分解动力学参数
Table 5. Kinetic parameters of thermal decomposition for H2L and 1
Compound β/(℃·min-1) Tp/℃ Kissinger Eq. Ozawa Eq. Ea/(kJ·mol-1) lgA r Ea/(kJ·mol-1) r H2L 5.00 291.50 10.00 303.55 145.8 11.15 -1.000 0 147.8 -1.000 0 15.00 310.89 1 5.00 290.69 10.00 297.84 264.1 22.39 -0.998 9 260.2 -0.999 0 15.00 301.41 -

 扫一扫看文章
扫一扫看文章
计量
- PDF下载量: 1
- 文章访问数: 635
- HTML全文浏览量: 101


















 下载:
下载:
















 下载:
下载:

