1 聊城大学药学院 聊城 252000;
2 聊城大学生物制药研究院 聊城 252000
2015-09-04 收稿, 2015-10-29 接受
基金项目: 山东省重点研发计划项目(2015GSF118160)资助
作者简介:
苏心怡, 女, 硕士, 从事抗肿瘤药物制剂研究
Study on Thermal Stability of Human Serum Albumin at Different pH Values and Its Structural Reversibility
1 School of Pharmacy, Liaocheng University, Liaocheng 252000;
2 Institute of BioPharmaceutical Research, Liaocheng University, Liaocheng 252000
人血清白蛋白(HSA)是血浆中的主要成分[1],可以维持人体内渗透压的平衡,并对许多内源性和外源性化合物进行储存和转运,对药物分子在体内的储存、转运和作用部位药理活性的发挥起着重要作用[2]。HSA由585个氨基酸组成,分子量为66500 Da。HSA的三维晶体结构表明其包含3个结构相似的功能域(domainⅠ, Ⅱ, Ⅲ)。每个功能域又包含2个结构相似的亚结构域(subdomain A, subdomain B),它的结构包括17个二硫键桥,分布在各个三级结构区域,同时还包括1个自由硫。许多研究结果证明,配体和药物的结合位点位于domainⅡA和domainⅢA[3, 4]。
白蛋白容易变性,其变性与其空间结构的改变有着密切关系。虽然白蛋白的氨基酸序列在其正确折叠中起着核心作用,但各种各样的因素,包括温度、pH、浓度、其他小分子对白蛋白的作用都会影响白蛋白的折叠,从而改变白蛋白的生理活性,为白蛋白生物制品及白蛋白药物的开发带来影响。临床药物的开发需要对不同白蛋白在不同状态下的空间结构和去折叠过程进行研究,以寻找在较大条件改变下仍能保持生理活性的蛋白,用于进一步的开发。Michnik等[5]研究了低浓度乙醇对HSA的影响,结果表明,随着乙醇浓度的增大,HSA的变性温度逐渐降低,即热稳定性变差。Shaw等[6]利用光谱学手段研究了温度对HSA稳定性的影响,结果表明,在25℃、60℃、75℃三个温度下,随着温度升高,HSA的α-螺旋结构比例逐渐降低。在pH 7.0条件下,白蛋白结构呈现正常的结构状态,即蛋白完全折叠;当pH低于4.3时,白蛋白结构会发生急剧的变化,部分去折叠;而逐渐减小到2.7时,白蛋白结构已处于完全伸展的状态;当pH高于8.0时,白蛋白结构处于中间状态,当pH高于10.0时,白蛋白结构变成了另外一种变性状态[1]。
由于HSA特殊的靶向性及载体特性,越来越多的人致力于药物和HSA作用的研究[7~9]。尽管血液pH通常较稳定,但是在药物与蛋白作用的细胞内外环境中,pH有较大改变,从而影响药物与蛋白作用。已有文献报道,pH对牛血清白蛋白(BSA)和HSA的结构影响[10, 11],但目前尚未见pH对HSA的结构可逆性影响的报道。差示扫描量热法(DSC)可以直接测量生物大分子内部结构的稳定性,并能提供准确的热变性温度(Tm)和热力学信息。动态光散射(dynamic light scattering, DLS)可以测量粒度大小,从而监测蛋白质的结构变化。圆二色谱(circular dichroism, CD)分析法可检测溶液中的蛋白二级结构的组成。本文利用DSC、DLS和CD研究了不同pH下HSA的热变性、粒径大小和二级结构的变化,并对pH的结构可逆性进行了探讨。
MicroCal VP-DSC差示扫描量热仪(Malvarn),并用MircoCalThermoVac对样品进行脱气。Zetasizer Nano ZSP纳米粒度电位仪(Malvarn),Jasco J-810圆二色谱仪,METTLER TOLEDO FE20 pH计。HSA由广东双林生物有限公司提供,使用过程中对其进行了透析。柠檬酸、磷酸氢二钠、盐酸、氢氧化钠等均为市售分析纯试剂。实验用水为超纯水(Millipore)。HSA溶液用不同pH的0.05mol/L的柠檬酸缓冲溶液配制。
用0.05mol/L pH 3.0、5.0、7.0、9.0和11.0的缓冲液配制1mg/mL的HSA溶液,以0.05mol/L条件下相同pH的缓冲液作为参比。进行样品检测前至少扫描3次buffer-buffer基线,消除金属记忆效应,扫描结果应重复性好。扫描速率60℃/h,加热范围为25℃~90℃。其结果由VP-DSC自带分析软件MicroCal Origin7.0进行分析。
将未进行热稳定性实验的pH 3.0、5.0、9.0、11.0条件下的1mg/mL的HSA分别用1mol/L NaOH溶液和1mol/L HCl溶液调至pH7.0,以相同条件的缓冲液作为参比,按上述实验条件进行检测。
用0.05mol/L pH 3.0、5.0、7.0、9.0和11.0的缓冲液配制1mg/mL的HSA溶液,用0.22μm微孔滤膜过滤后置于Zetasizer Nano ZSP中进行检测,检测温度为25℃。分别将不同pH下1mg/mL的HSA调回pH7.0,进行粒度检测,每个样品平行测定3次。
用0.05mol/L pH 3.0、5.0、7.0、9.0和11.0的缓冲液配制0.2mg/mL的HSA溶液,以0.05mol/L条件下相同pH的缓冲液作为参比,进行CD实验。实验时光源系统用氮气保护(流量为5L/min),样品池光径为0.1cm。样品扫描速率为100nm/min,分辨率为0.1nm,响应时间1s,扫描波段为200~260 nm,每个样品累积扫描3次。分别将不同pH下0.2mg/mL的HSA调回pH 7.0,以相应缓冲液作为参比,按上述条件进行测定。
DSC法是一种直接鉴定蛋白质或其他生物大分子在自然状态下稳定性的技术。该技术通过测量与分子恒速加热时热变性相关的热量变化予以实现。溶液中的生物分子在其自然(折叠)和变性(展开)构象之间保持平衡。Tm值越高,分子就越稳定。DSC可以测量由热变性造成的去折叠热焓(ΔH),它也被用于确定变性过程中的热熔变化(ΔCp)。DSC的测量有助于说明对生物大分子折叠和保持稳定的因素,这些因素包括疏水相互作用、氢键结合和物理环境等,是一种非常高效的生物大分子结构变化检测手段[12, 13]。对蛋白质进行检测时,DSC可以用来确定蛋白质的热稳定性和折叠机制,同时也可以用来进行更多定性的筛选,例如,作为配体结合、制剂处方筛选、晶体生长条件选择的指标。热谱图中的Tm值反应蛋白的热变性,是蛋白到达不可逆温度的标志[14, 15]。
实验中分别测量了1mg/mL HSA在pH 3.0~11.0范围内的DSC谱图以比较其变化,结果见图 1。在pH 7.0条件下的HSA在68.17℃时出现了1个吸热峰;在pH 5.0条件下,HSA已经部分去折叠;当pH降到3.0时没有出现吸热峰,表明HSA高度有序的三级结构在加热前已被破坏。当HSA处于碱性环境时,pH 9.0条件下HSA出现了2个重叠峰,且峰形不明显;pH 11.0时HSA已无吸热峰,此时HSA的三级结构已改变或遭到破坏。
为了检测HSA的结构对不同pH条件是否具有可逆性,我们把未进行热稳定性实验的不同pH下的1mg/mL的HSA调回了7.0对其进行检测,结果见图 2。由图可见,在不同pH下三级结构已改变的HSA在pH调回7.0后的DSC检测中又出现了吸热峰,且Tm值与pH 7.0条件下的Tm值接近,酸性条件下的HSA调回pH 7.0后的Tm值略高于中性和碱性条件下HSA调回pH7.0后的Tm值。根据热力学原理,
|
$\Delta H=\int \Delta {{C}_{p}}dT$
|
(1)
|
检测过程中得出的不同pH条件下HSA的焓值结果见表 1。pH 5.0和7.0的焓值最大且基本相等,说明此区间内HSA的结构高度有序,其结构进行热转变时需要更多的能量。pH从5.0降低到3.0,焓值降低,说明HSA经过pH 3.0条件处理再调回pH 7.0,结构会重新进行折叠,但不能回到原始状态。同样,pH从9.0升高到11.0,焓值也发生了降低。对比pH 3.0和pH 11.0的HSA调回pH 7.0的焓值,碱性条件下的HSA回到中性状态的结构更加高度有序。
表 1
Table 1
表 1(Table 1)
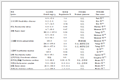
表 1 不同pH下HSA(1mg/mL)调回pH7.0后热力学参数
Table 1 Thermodynamic parameters of HSA(1mg/mL) when different pH values were adjusted back to 7.0
| pH | Tm/℃ | ΔH/(kcal·mol-1) |
| 3.0 | 69.25±0.11 | 154.1±3.2 |
| 5.0 | 68.96±0.06 | 196.8±2.8 |
| 7.0 | 68.17±0.06 | 196.6±2.8 |
| 9.0 | 67.47±0.08 | 179.4±3.0 |
| 11.0 | 67.53±0.07 | 168.1±2.3 |
|
表 1 不同pH下HSA(1mg/mL)调回pH7.0后热力学参数
Table 1 Thermodynamic parameters of HSA(1mg/mL) when different pH values were adjusted back to 7.0
|
DLS技术可以测量亚微细颗粒范围内的分子与颗粒的粒度及粒度分布,其典型应用包括已分散或溶于液体的颗粒、乳剂等的分子表征。悬浮在溶液中的颗粒的布朗运动,造成散射光光强的波动。对于DLS来说,布朗运动的一个重要特点是小分子运动快速,大分子运动缓慢。在斯托克斯-爱因斯坦方程中,定义了粒径与其布朗运动所致速度之间的关系[16, 17]。Zetasizer Nano ZSP可以通过测量光强波动的速度,然后用于计算粒径。
在酸性和碱性条件下,HSA的结构发生了去折叠,此时蛋白内部共价键断裂,结构伸展,从而粒径也发生了改变。我们从实验中得出了不同pH下HSA的粒径及其分布(图 3)。在pH 7.0的条件下,HSA的粒径为9.68nm,此时白蛋白的结构处于正常状态。当pH从5.0降到3.0的过程中,粒径急剧变大,从10.05nm增加到了 15.63nm。这也解释了HSA的结构在此pH区间发生了急剧的去折叠。同样,在pH由9.0到11.0的过程中,粒径由12.33nm增加到了19.60nm。碱性条件下的HSA的伸展更大,其粒径变化趋势见图 4。当我们把在不同pH条件下发生去折叠的HSA调回pH 7.0时,粒径减小,但不能恢复到中性条件下的HSA的粒径(图 5)。因此可以得出不同pH下的HSA调回中性时,HSA的粒径有接近于中性条件下HSA粒径的趋势。
HSA有光学活性,CD可以测定其立体结构。根据CD谱(图 6,图 7)的研究结果可以看出,在不同pH条件下,HSA的二级结构单元含量是不同的。由CD实验结果求得HSA中α-螺旋含量的计算公式为
|
$\text{MRE=}\frac{\text{ObservedCD}\left( \text{mdegree} \right)}{{{C}_{p}}nl\times 10}$
|
(2)
|
|
${\rm{\alpha - Helix}}\left( {\rm{\% }} \right){\rm{ = }}\frac{{{\rm{MR}}{{\rm{E}}_{{\rm{208}}}} - 4000}}{{33000 - 4000}} \times 100$
|
(3)
|
其中,Cp是蛋白的摩尔浓度,n为氨基酸残基的数量,l为样品池光径。pH 7.0条件下HSA中α-螺旋含量为29.8%,pH 3.0、5.0、9.0和11.0条件下的HSA的α-螺旋含量分别为41.2%、37.8%、30.2%和47.3%。说明pH改变,HSA的二级结构也会发生相应改变。当不同pH下的HSA调回pH 7.0时,pH 3.0、5.0、9.0和11.0的HSA中α-螺旋含量分别为41.5%、44.5%、51.6%和59.3%。说明不同pH下的HSA在调回pH 7.0时结构会发生改变,但二级结构不能恢复到中性条件下的状态。
DSC法对不同pH下的HSA的热力学研究结果表明,酸性和碱性条件下,HSA的三级结构会发生不同程度的去折叠。当pH调回中性时,去折叠的结构又重新进行了折叠,但无法恢复至中性pH下HSA的结构状态。变性温度和正常结构时的变性温度基本相同。DLS实验通过表征HSA粒径大小,说明了在酸性和碱性条件下HSA的粒径变大,HSA的结构发生了去折叠,且酸(碱)性越大,去折叠程度越大。将pH调回7.0时,HSA的粒径又开始减小,说明此时HSA结构又经历了折叠过程。CD实验说明了pH的改变会影响HSA的二级结构,当不同pH条件下的HSA调回pH 7.0时,二级结构会发生改变,但不能恢复到HSA在中性条件时的结构。结合以上3个实验,表明HSA结构在不同pH下调回中性条件时结构会重新折叠,但不能恢复至中性条件下的结构状态。pH的改变导致HSA的结构转变,有可能暴露出更多的疏水结构,这对指导难溶性小分子化合物与HSA的结合有着重要意义。
 2016,
Vol. 79
2016,
Vol. 79 Issue (4): 344-348
Issue (4): 344-348 2016,
Vol. 79
2016,
Vol. 79 Issue (4): 344-348
Issue (4): 344-348