2 清华大学化学系, 北京 100084
2 Department of Chemistry, Tsinghua University, Beijing 100084, China
微流控芯片的概念于20世纪90年代初由Manz等提出,是指在几平方厘米的芯片上构建的化学或生物实验室,实现化学和生物学等研究领域中的样品制备、反应、分离、检测和细胞培养、分选等基本功能。人类目前对于生命的本质和规律认识欠缺,众多科研人员投身到基因组学、代谢组学、蛋白质组学等生物研究中,体外研究细胞、组织或器官的需求极大地促进了微流控技术用于生物应用的发展,微流控技术广泛应用于药物筛选[1, 2]、细胞分析[3]、细胞捕获[4]、生物小分子(如核酸、蛋白质等)分析[5]等生命分析化学领域。与上述生物应用息息相关的微流控技术主要有:芯片材料与制作技术、表面改性技术、样品前处理技术、液滴技术和检测等。微流控芯片材料的选取决定芯片制作方法和通道内壁的表面性质,如常见的聚二甲基硅氧烷(PDMS)芯片需采用标准软光刻技术制作,通道内壁为疏水表面;而以滤纸为材料的纸芯片[6]制作常用喷蜡打印法,且滤纸本身具有良好的亲水性。表面改性技术是对选取的芯片材料不足的补充,通过对通道内壁进行功能化修饰,使通道内壁表现出更理想的表面性质。He等[7]采用浸润的方法将2% PVA-5% Gly固定在PDMS芯片通道表面,使原本疏水的通道表面变成超亲水的表面。对芯片中微流体实现精确而灵活的操控是微流控芯片应用的基础。在微流控芯片的微米级通道内,由于通道几何结构的限制,两相流体保持平行流动[8],通过对通道几何结构的优化以及两相流体的组成和流速等条件的控制可以使其保持层流或生成微液滴。液滴技术的关键是液滴的生成与操控,本课题组开展了铁磁流体液滴的研究工作,以铁磁流体为连续相制备微液滴,实现微流控芯片上二维液滴的操控,包括控制液滴的尺寸、数量和液滴的移动,并且对磁操控液滴的原理和技术进行了系统的研究,拓展了该技术在化学生物学研究中的应用[9~12]。液滴技术具有液滴生成速度快、通量高[13]等独特的优势,可作为载体用于细胞分选[14]、细胞培养[15]、单细胞捕获[16]和功能性粒子合成[17]等方面。
本文以涉及细胞的微流控技术为切入点,重点介绍了近五年来芯片材料与制作技术、表面改性技术和液滴技术的发展,以及微流控技术在药物筛选和细胞分析(包括单细胞分析、细胞培养、组织或器官模拟)等方面的应用进展。
2 微流控技术微流控芯片是芯片实验室的核心,涉及芯片材料与制作、表面改性、微流体的操控等技术,其中芯片材料与制作技术和表面改性技术均对芯片通道内壁的表面性质有巨大的影响,研究过程中需根据实际情况选择合适的芯片材料和修饰方法。此外,液滴技术是在微流控芯片上发展起来的一种新的操作微小体积液滴的技术,在药物筛选和细胞分析方面得到了较为广泛的应用。微流控液滴技术具有(1)液滴体积小、比表面积大;(2)生成速度快、通量高;(3)液滴尺寸均匀、分散性小;(4)液滴内体系封闭、无交叉污染等优点,是微观尺度上研究细胞的重要平台。
2.1 芯片材料与制作技术基于细胞的微流控芯片常用的制作材料主要有PDMS[18]、聚甲基丙烯酸甲酯(PMMA)[19]以及水凝胶等。PDMS和PMMA是实验室中常见的芯片材料,均属于聚合物材料。PDMS自身具有良好的透气性、透光性和生物相容性,易加工成型,成本低,应用最为广泛,但耐压性差、易形变等特点限制了PDMS在某些分离芯片上的应用,却为其应用于气动阀等制作提供了可能。与PDMS相比,PMMA制作的芯片硬度更大,封接后的芯片更牢固,能承受较大的压力,然而PMMA芯片制作过程相对繁琐。水凝胶是一种高分子网状体系,所具有的高分子网络结构,能为三维培养细胞提供三维生长的骨架[20],因此,芯片上的三维细胞培养系统一般需在细胞培养室中注入水凝胶和细胞的混合溶液[21]。除上述材料外,研究人员也将聚碳酸酯(PC)[22]等高聚物用作芯片制作材料,进行细胞分析和药物筛选等相关研究。PC材料属于热固性材料,质地坚硬,冲击强度极佳,但PC芯片的制作工艺复杂。Churski等[23]以聚碳酸酯为芯片材料,用数控铣机器制作,结合微流控液滴装置用于检测氨苄青霉素、四环素、氯霉素对大肠杆菌的最小抑制浓度及抗生素间的相互作用。纸芯片微流控技术是一种新型微流控技术,与传统的微流控芯片相比,纸芯片材料来源丰富、成本低,试样消耗更少,生物相容性好,依靠毛细管作用驱动流体而不用外加注射泵。二维纸芯片常用的制作方法为喷蜡打印法。纸芯片的独特优势,使其在细胞培养和药物筛选[24]等方面得到应用。近年来,3D打印技术不断发展,使用3D打印技术制作微装置,操作简单方便,能够直接添加制作材料,而不需要额外的刻蚀或溶解处理,制作过程环保、效率高[25]。然而,3D打印技术也存在加工材料种类有限,打印精度较低,表面平整度不佳等问题,限制了3D打印在微流控技术中的推广和应用。目前,3D打印技术已用于打印制作芯片器官[26]、类皮肤软组织[27]和三维微血管网络[28]等,弥补了传统芯片加工技术难以制作复杂空间结构上的不足,使体外仿生模型更立体、更真实。
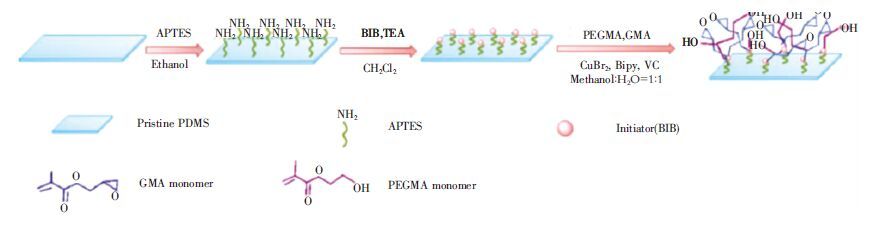
2.2 表面改性技术芯片材料的表面化学性质对细胞分析具有较大的影响。对细胞尤其是贴壁细胞来说,细胞成功贴壁后才能在通道表面进行后续细胞的扩散、转移和增殖等过程,因此,有必要对通道内壁进行改性处理,创造适合细胞生存的微环境。以PDMS微流控芯片为例,PDMS芯片通道内壁表现为疏水性,对细胞的粘附力较差。 Yu等[29]通过表面引发的原子转移自由基聚合法(如图 1)在PDMS的表面修饰上一种较复杂的高聚物,引入环氧基、羟基等亲水基团,改善了细胞生长的贴壁能力。研究表明,采用聚L-赖氨酸或聚D-赖氨酸涂层[30]的方法也可以提高细胞的贴壁能力。此外,Yoshimitsu等[31]还研究了不同类型细胞外基质修饰通道内壁对细胞生存的影响,结果发现,在纤连蛋白、层连蛋白、胶原和明胶四种基质中,纤连蛋白和层粘连蛋白修饰的PDMS表面培养的多能干细胞的生存状态更好。并不是所有的研究都需要细胞贴壁生长,对于三维培养肿瘤微球的研究来说,需要防止细胞的贴壁生长,而采用PVA修饰PDMS 表面的方法[32, 33]可以很好地阻止细胞在芯片表面的粘附。
|
图 1 聚合物功能化修饰PDMS的过程[29] Figure 1 Poly(GMA-co-pEGMA)-functionalized PDMS[29] APTES: 3-amminopropyl triethoxysilane; BIB: α-bromoisobutyryl bromide; TEA: triethylamine; GMA: glycidyl methacrylate; PEGMA: poly(ethylene glycol) methacrylate; Bipy: 2,2-bipyridyl; VC: L-ascorbic acid. |
2.3 液滴技术
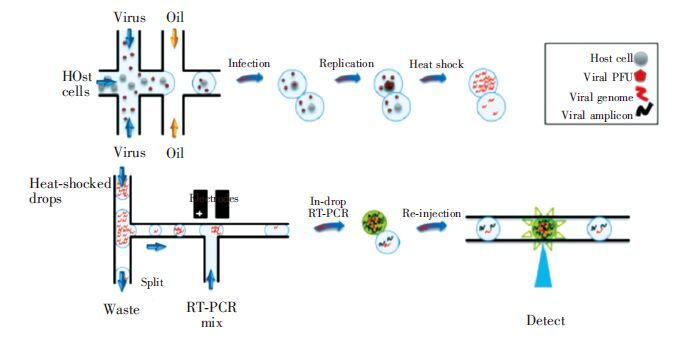
在微流控装置中,将两相互不相溶的流体,一种作为连续相,另一种作为分散相,分散相在连续相的剪切力作用下,以微小体积的形式分散于连续相中,这一过程即为微液滴的生成。液滴技术所生成的微液滴体积小,一般为纳升级甚至皮升级,与细胞的尺寸接近,小分子或细胞等可被封装在单分散的液滴内形成独立的分析系统。液滴技术制备的微液滴可作为微反应器、且液滴内与外界无交叉污染,可用作细胞捕获[34]、细胞分选等的平台。El Debs等[35]成功地利用液滴微流控为平台来分选杂种瘤细胞,使其释放能抑制临床相关药物靶标的抗体,分选速率达到5万个细胞/小时。该方法将单个细胞直接封装在水相液滴中,然后进行荧光检测抑制药物靶标的抗体的释放。同时,液滴技术也可用于体外金属纳米粒子的生物合成的研究[36]。Tao等[37]巧妙地利用液滴的融合(实验示意图如图 2所示),将感染型病毒与宿主细胞一起培养在皮升级液滴中,开展病毒感染性的快速研究。在上述液滴技术相关的研究中,均以油相为连续相,以细胞悬浮液为分散相,生成油包水的液滴。与上述研究不同的是,Sabhachandani等[38]以海藻酸水溶液为分散相,制备出单分散的负载细胞的海藻酸水凝胶液滴,肿瘤细胞依靠水凝胶的分子骨架最终培养出三维肿瘤微球。随着3D打印技术的风靡,Villar等[39]以3D生物打印的方法为基础,开发了一种合成类生物组织材料的打印方法。他们利用该方法打印出数以万计的液滴,液滴之间由于单脂质双层的存在最终形成紧密连接、协同操作的液滴网络结构,这种网络结构可用膜蛋白功能化修饰,从而允许电信号沿着特定路径的快速传播,并且这一网络还可通过渗透压梯度进行自主调控,使其按照可预测的方式折叠形成直接打印难以实现的结构。
3 微流控技术的应用
细胞是生物体的基本结构和功能单元。细胞分析主要研究细胞行为,包括细胞的生长、增殖、分化、入侵、转移和凋亡等生理过程。细胞分析对于认识生命体发展和基本规律、药物作用机制和细胞毒性、疾病早期诊断和治疗等都是非常必要的。微流控技术所具有的样品和试剂用量小、高通量分析的特点,在生物应用方面表现出巨大的应用潜能。细胞的微流控技术的应用主要分为细胞分析、药物筛选和核酸分析等,其中在细胞分析方面主要介绍单细胞分析、细胞培养和体外组织或器官模拟的研究进展。
3.1 细胞分析 3.1.1 单细胞分析细胞的尺寸极小,由于检测器灵敏度的限制,人们只能从细胞群的角度来进行生命分析。对细胞群的研究只能获得统计的平均结果,忽略了细胞间的个体差异,而单细胞分析能够帮助我们从细胞水平上理解复杂的生物系统,单细胞分析已成为近年来的研究热点。在单细胞分析中,最重要的是实现单细胞的分离与捕获,在形成单细胞后才能根据实际需求进行后续的单细胞分析。目前,微流控芯片上单细胞的捕获方式主要有液滴捕获[40, 41]和基于各种微结构的单细胞阵列捕获[42]。液滴微流控芯片上快速生成的单分散微液滴可作为高灵敏度单细胞分析的微反应器,液滴捕获单细胞的方法就是将每个细胞分别包裹在离散的水相液滴中。Li等[43]采用液滴捕获单细胞的方法,在适配体和切断酶的辅助作用下,利用荧光检测法成功检测到活体单细胞表面的膜蛋白。液滴捕获的方法通量高,但需要经过充分的条件优化才能达到一定的捕获效率,对于易聚集的细胞,不能保证每个液滴均能捕获到细胞以及捕获到的是单个细胞。单细胞阵列捕获细胞的原理是,在芯片通道内设计与单个细胞尺寸相匹配的微井[44]、U型微栅栏[45]等结构,在通道内通入细胞悬浮液时,利用通道内的分布微型结构,使细胞保留在这些微结构中,从而获得单个细胞。这种方法对芯片的制作有更高的要求,微井或微栅栏的尺寸要与细胞尺寸相匹配。Shih等[41]综合以上两种细胞捕获的方法,首先用液滴微流控技术生成包裹细胞的微液滴,然后微液滴分别进入阵列分布的通道内形成负载细胞的微液滴阵列,最后利用该方法研究了药物敏感型和耐药型两种乳腺癌细胞对药物吸收和代谢、细胞毒性的动力学。
3.1.2 细胞培养细胞培养是生物应用的基础,微流控芯片上的细胞培养可分为二维细胞培养、三维细胞培养和三维共培养。与传统的96-孔板或培养皿上的培养系统不同,微流控芯片上的细胞培养可采用动态灌注培养[46, 47],此方法培养的细胞具有比静态培养更多的功能。 Matharu等[48]在微流控芯片上进行肝星状细胞的连续灌注培养,实现了活化的肝星状细胞中转化生长因子释放的实时监测。微流控芯片上三维细胞的培养[49]更接近体内的真实环境,是目前的一个热门研究领域,尤其是三维肿瘤微球的培养[50, 51]研究最多,三维肿瘤微球模型可用于抗癌药物筛选和肿瘤转移的研究。三维细胞共培养是指在一个芯片上同时培养两种或多种不同的细胞,是体外器官模拟的基础,可以构建肿瘤细胞生长[52]或血管再生[53]等的微环境。Zhang等[54]曾建立了一种简易的体外肝脏模型,将HepG2和MCF-7细胞(其中HepG2代表肝脏,MCF-7代表肿瘤细胞)分别置于两个相互连通的微通道中共培养,保证细胞间有效的物质交换,并结合质谱检测器证明了卡培他滨(CAP)首先在肝脏中代谢为中间产物后,再发挥治疗肿瘤的功效。与之不同的是,van der Meer等[55]则直接在一个微通道中注入人类脐静脉内皮细胞、人胚胎干细胞衍生的周皮细胞(血管平滑肌细胞的前体)和鼠尾胶原I的混合物,通过内皮细胞和周皮细胞的三维共培养来观察体外血管组织的三维构建。芯片上细胞培养方法的不断改进,使得体外模型细胞培养的微环境与体内状态尽可能的相近,为芯片上的体外组织和器官模拟奠定了基础。
3.1.3 体外组织或器官模拟体外组织或器官模拟的目标是建立尽可能接近体内真实环境的仿生体系,考虑细胞-细胞/蛋白等的相互作用的影响,帮助人们体外研究生命体的本质和规律,因此,芯片上的组织或器官模拟提供了一种相对简单的研究生物体的途径。血管是药物运输的重要途径,建立可靠的体外微血管模型是组织工程的重要目标。因此,用微流控系统体外模拟三维微血管网络[56, 57]的研究成果较多,Chen等[58]开发了一个双层膜结构的微流控装置来模拟血管-瓣膜三维微环境,以明胶甲基丙烯酸酯水凝胶作为内皮下生理相关的三维基质,根据生理环境合理安排多种细胞类型的空间分布,准确控制内皮单细胞层的流体流动速率并采用多孔膜来维持细胞的划分而允许异性细胞间的相互作用,这一系列实验条件的控制使体外模型更接近体内的生理环境;Lee等[59]用微流控平台模拟血管网络结构组建了检测血管渗透性的模型,验证了血管的屏障功能。同时,微流控芯片上可模拟的组织或器官有骨组织[60]、心脏[61]、皮肤[62]以及肝脏-皮肤共培养的多器官芯片[63]等。除了简单模拟体内微环境之外,微流控装置上也可实现器官功能的模拟,Huh等[64]建立了一个多功能微装置,模拟肺部的呼吸功能,进行体外重构肺泡-毛细管屏障的微体系结构,结果表明,肺模拟装置对引入到肺泡-毛细管屏障上的细菌和炎症性细胞因子表现出器官水平的复杂响应。这一研究在模拟器官基本构造的基础上更进一步,实现了器官功能的模拟,推动了器官芯片技术的继续发展。阿兹海默症等神经退行性疾病是当今老龄化社会普遍存在的疾病,体外脑芯片模型[65]有助于人类深入了解此类疾病的病因,血脑屏障的体外模型[66]为神经退行性疾病的治疗创造了条件。此外,研究者们对乳腺癌[67, 68]等疾病的体外模型也做了相关的研究,对临床上乳腺癌的早期发现和治疗有重要意义。
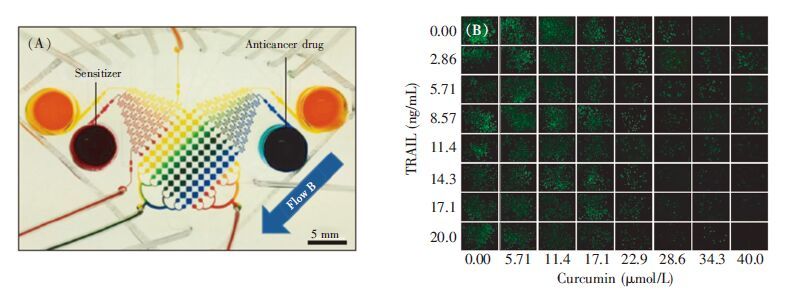
3.2 药物筛选新药研发的一个关键步骤是药物筛选,即对可能作为药物使用的物质进行生物活性、药理作用及药用价值的评估。传统的药物筛选方法样品消耗大、分析时间长、通量低,基于微流控技术的药物筛选方法弥补了传统方法的不足。最简单的药物筛选微流控芯片是以二维培养的细胞为基础,可根据不同药物对细胞生存[69, 70]的作用进行筛选,也有利用细胞的趋药性[71, 72]研究药物对细胞生长的影响,同时,单细胞水平的药物筛选[73, 74]也被证实可行。但是,这些二维细胞培养的方法操作复杂,一次只能分析一种参数的某种药物,筛选的通量较低。于是,利用细胞阵列的微流控芯片的组合药物筛选方法逐步发展[75, 76],这种方法能够在微流控芯片上自动生成浓度梯度,同时进行多种药物浓度或联合用药的高通量筛选[77]。An等[78]设计了一种高通量药物筛选系统,芯片上自动生成两种参数梯度,如图 3所示,同时分析姜黄素/肿瘤坏死因子α相关的细胞凋亡诱导配体的组合化学疗法对人前列腺肿瘤细胞的作用效果。三维细胞培养是比二维细胞培养更接近体内细胞生存的体系,因此,基于三维细胞培养的药物筛选系统可信度更高。根据细胞种类不同,以三维细胞培养为基础的药物筛选又可分为一种细胞三维培养[79, 80]和两种细胞共培养[52]两种筛选类型。Gao等[81]构建了一个集成浓度梯度发生器、细胞培养室、固相萃取柱于一体的微流控装置,研究了氨甲喋呤(MTX)对三维共培养的人肝癌细胞和人结直肠癌细胞的影响,并用ESI-Q-TOF-MS进行检测,证明了药物吸收的特性与在线质谱分析同时进行的可行性。
|
图 3 芯片通道内浓度梯度生成与细胞分析[78]。(A) 芯片通道内通入不同颜色的染料溶液演示浓度梯度的生成;(B) 组合药物作用后培养24 h的人前列腺癌细胞的荧光显微镜图。 Figure 3 Generation of concentration gradient in microfluidic and cell analysis[78]. (A) Demonstration of concentration gradient in microfluidic system using color dye solution; (B) Representative fluorescent micrographs of PC3 cells after sequential treatment with curcumin and TRAIL for 24 hours each. |
3.3 核酸分析
早期微流控芯片主要由毛细管电泳技术演变而来,芯片电泳[82]可用于DNA的萃取和富集,有效提高了DNA的分离效率。Reed等[83]将微流控芯片与阵列结合,在微通道内引入微型固相萃取柱,该装置能完成复杂生物样本(全血)中的DNA分离,纯化后的DNA可直接用作PCR扩增。与微阵列类似,Abate等[84]发展了基于液滴微流控的DNA序列分析方法,结合FRET连接反应直接分析DNA序列,明显增加了DNA分析的通量。PCR反应是基因诊断中的核心技术,液滴数字PCR[85]已有商业化产品,能做到样品中靶标核酸的精确定量。黄岩谊及其同事[86]集成液滴技术和PCR反应,开发了单细胞基因组的乳液全基因组扩增技术,显著提高了扩增的均匀性和准确度,该方法首次实现了人体单细胞中基因组拷贝数变异和单核苷酸变异的同时检测。已有报道集成的DNA分析芯片[87, 88]能完成样品制备到毛细管电泳分析的完整流程,做到"样品进-结果出"的快速分析。在整个分析流程中,集成的装置能精确控制芯片上PCR反应的温度,降低总体分析时间,大幅减少试剂消耗,相信高度集成的分析装置将成为未来DNA分析的主流。
4 总结与展望微流控技术在生命分析化学领域得到了巨大的发展,研究人员对生物体本质和规律的研究热情,极大地促进了微流控技术及其在细胞水平应用的研究,包括单细胞、亚细胞到多细胞(组织器官)水平的研究。当前,微流控分析技术的发展主要面临以下几个问题:(1)生物科学的研究对芯片制作材料的表面性质、机械性能和生物相容性等提出了更高的要求,针对特定的研究需求往往要求某方面特殊性能或者是兼具多种性能的材料;(2)目前微流控芯片的检测技术以光学检测为主,电化学检测和质谱检测有一些新的进展,但是微流控芯片检测技术仍然是一个发展瓶颈,而且检测器与芯片的集成化依然进展缓慢;(3)目前微流控技术仍主要停留在实验室阶段,芯片分析技术的稳定性、可重复性有待改进,芯片设计和制作过于强调订制化而缺少标准化、规模化,便携、集成化的微流控芯片仪器和配套装置亟待发展,这些都制约了微流控技术的产业化发展。当今社会,人类对生命科学本质的认识和对健康的追求,极大地促进了微流控技术在单细胞分析和器官模拟等生命科学领域的应用,单细胞分析和器官芯片将是未来微流控技术发展的研究热点。虽然现阶段的研究,微流控芯片构建的体外模型与真实生理环境仍存在较大的差距,相信未来器官芯片将会兼顾细胞生存的微环境和器官功能等多方面因素的影响,结合更多新材料和新型检测技术的引入,实现更复杂、更真实的模拟人体的研究,辅助基因组学、蛋白质组学、代谢组学等研究,促进个性化诊疗和精准医疗的发展。
| 1 |
Ma W Y, Hsiung L C, Wang C H, Chiang C L, Lin C H, Huang C S, Wo A M. Sci. Rep., 2015, 5 :9656 doi:10.1038/srep09656 |
| 2 |
Caviglia C, Zor K, Montini L, Tilli V, Canepa S, Melander F, Muhammad H B, Carminati M, Ferrari G, Raiteri R, Heiskanen A, Andresen T L, Emneus J. Anal. Chem., 2015, 87 (4):2204–2212 doi:10.1021/ac503621d |
| 3 |
Compton J L, Luo J C, Ma H, Botvinick E, Venugopalan V. Nat. Photonics, 2014, 8 :710–715 doi:10.1038/nphoton.2014.165 |
| 4 |
Liu Z, Zhang W, Huang F, Feng H, Shu W, Xu X, Chen Y. Biosens. Bioelectron., 2013, 47 :113–119 doi:10.1016/j.bios.2013.03.017 |
| 5 |
Lai D, Frampton J P, Tsuei M, Kao A, Takayama S. Anal. Chem., 2014, 86 (8):4052–4057 doi:10.1021/ac500657k |
| 6 |
Tao F F, Xiao X, Lei K F, Lee I C. Biochip J, 2015, 9 (2):97–104 doi:10.1007/s13206-015-9202-7 |
| 7 |
He T, Liang Q, Zhang K, Mu X, Luo T, Wang Y, Luo G. Microfluid. Nanofluid., 2011, 10 (6):1289–1298 doi:10.1007/s10404-010-0760-7 |
| 8 |
Mu X, Liang Q, Zhou J, Ren K, Hu P, Wang Y, Zheng Z, Luo G. Biomed. Microdevices, 2014, 16 (2):245–253 doi:10.1007/s10544-013-9828-y |
| 9 |
Zhang K, Liang Q, Ma S, Mu X, Hu P, Wang Y, Luo G. Lab Chip, 2009, 9 (20):2992–2999 doi:10.1039/b906229g |
| 10 |
Zhang K, Liang Q, Ai X, Hu P, Wang Y, Luo G. Anal. Chem., 2011, 83 (20):8029–8034 doi:10.1021/ac2017458 |
| 11 |
Zhang K, Liang Q, Ai X, Hu P, Wang Y, Luo G. Lab Chip, 2011, 11 (7):1271–1275 doi:10.1039/c0lc00484g |
| 12 |
Pamme N. Curr. Opin. Chem. Biol., 2012, 16 (3-4):436–443 doi:10.1016/j.cbpa.2012.05.181 |
| 13 |
Conchouso D, Castro D, Khan S A, Foulds I G. Lab Chip, 2014, 14 (16):3011–3020 doi:10.1039/C4LC00379A |
| 14 |
Lee C, Lee J, Kim H H, Teh S Y, Lee A, Chung I Y, Park J Y, Shung K K. Lab Chip, 2012, 12 (15):2736–2742 doi:10.1039/c2lc21123h |
| 15 |
Wang Y, Wang J. Analyst, 2014, 139 (10):2449–2458 doi:10.1039/C4AN00015C |
| 16 |
Ying D, Zhang K, Li N, Ai X, Liang Q, Wang Y, Luo G. Biochip. J., 2012, 6 (3):197–205 doi:10.1007/s13206-012-6301-6 |
| 17 |
Han D J, Jung J H, Choi J S, Kim Y T, Seo T S. Lab Chip, 2013, 13 (20):4006–4010 doi:10.1039/c3lc50838b |
| 18 |
Kim S, Chung M, Jeon N L. Biomaterials, 2016, 78 :115–128 doi:10.1016/j.biomaterials.2015.11.019 |
| 19 |
Weltin A, Slotwinski K, Kieninger J, Moser I, Jobst G, Wego M, Ehret R, Urban G A. Lab Chip, 2014, 14 (1):138–146 doi:10.1039/C3LC50759A |
| 20 |
Tocchio A, Martello F, Tamplenizza M, Rossi E, Gerges I, Milani P, Lenardi C. Acta Biomater., 2015, 18 :144–154 doi:10.1016/j.actbio.2015.02.017 |
| 21 |
Mu X, Zheng W, Xiao L, Zhang W, Jiang X. Lab Chip, 2013, 13 (8):1612–1618 doi:10.1039/c3lc41342j |
| 22 |
Chen Q, He Z, Liu W, Lin X, Wu J, Li H, Lin J-M. Adv. Healthc Mater., 2015, 4 (15):2291–2296 doi:10.1002/adhm.201500383 |
| 23 |
Churski K, Kaminski T S, Jakiela S, Kamysz W, Baranska-Rybak W, Weibel D B, Garstecki P. Lab Chip, 2012, 12 (9):1629–1637 doi:10.1039/c2lc21284f |
| 24 |
Su M, Ge L, Ge S, Li N, Yu J, Yan M, Huang J. Anal. Chim. Acta, 2014, 847 :1–9 doi:10.1016/j.aca.2014.08.013 |
| 25 |
Au A K, Huynh W, Horowitz L F, Folch A. Angew. Chem. Int. Ed. Engl., 2016, 55 (12):3862–3881 doi:10.1002/anie.201504382 |
| 26 |
Bhise N S, Manoharan V, Massa S, Tamayol A, Ghaderi M, Miscuglio M, Lang Q, Shrike Zhang Y, Shin S R, Calzone G, Annabi N, Shupe T D, Bishop C E, Atala A, Dokmeci M R, Khademhosseini A. Biofabrication, 2016, 8 (1):014101 doi:10.1088/1758-5090/8/1/014101 |
| 27 |
Rimann M, Bono E, Annaheim H, Bleisch M, Graf-Hausner U. J. Lab. Autom., 2016, 21 (4):496–509 doi:10.1177/2211068214567146 |
| 28 |
Wu W, DeConinck A, Lewis J A. Adv. Mater., 2011, 23 (24):H178–183 doi:10.1002/adma.201004625 |
| 29 |
Yu L, Shi Z, Gao L, Li C. J. Biomed. Mater Res. A, 2015, 103 (9):2987–2997 doi:10.1002/jbm.a.v103.9 |
| 30 |
Tong Z, Segura-Feliu M, Seira O, Homs-Corbera A, Del Río J A, Samitier J. RSC Adv., 2015, 5 (90):73457–73466 doi:10.1039/C5RA11522A |
| 31 |
Yoshimitsu R, Hattori K, Sugiura S, Kondo Y, Yamada R, Tachikawa S, Satoh T, Kurisaki A, Ohnuma K, Asashima M, Kanamori T. Biotechnol. Bioeng., 2014, 111 (5):937–947 doi:10.1002/bit.25150 |
| 32 |
Kwapiszewska K, Michalczuk A, Rybka M, Kwapiszewski R, Brzozka Z. Lab Chip, 2014, 14 (12):2096–2104 doi:10.1039/C4LC00291A |
| 33 |
Chen Y, Gao D, Liu H, Lin S, Jiang Y. Anal. Chim. Acta, 2015, 898 :85–92 doi:10.1016/j.aca.2015.10.006 |
| 34 |
Platzman I, Janiesch J W, Spatz J P. J. Am. Chem. Soc., 2013, 135 (9):3339–3342 doi:10.1021/ja311588c |
| 35 |
El Debs B, Utharala R, Balyasnikova I V, Griffiths A D, Merten C A. P. Natl. Acad. Sci. USA, 2012, 109 (29):11570–11575 doi:10.1073/pnas.1204514109 |
| 36 |
Juul S, Nielsen C J, Labouriau R, Roy A, Tesauro C, Jensen P W, Harmsen C, Kristoffersen E L, Chiu Y L, Frohlich R, Fiorani P, Cox-Singh J, Tordrup D, Koch J, Bienvenu A L, Desideri A, Picot S, Petersen E, Leong K W, Ho Y P, Stougaard M, Knudsen B R. ACS nano, 2012, 6 (12):10676–10683 doi:10.1021/nn3038594 |
| 37 |
Tao Y, Rotem A, Zhang H, Chang C B, Basu A, Kolawole A O, Koehler S A, Ren Y, Lin J S, Pipas J M, Feldman A B, Wobus C E, Weitz D A. Lab Chip, 2015, 15 (19):3934–3940 doi:10.1039/C5LC00556F |
| 38 |
Sabhachandani P, Motwani V, Cohen N, Sarkar S, Torchilin V, Konry T. Lab Chip, 2016, 16 (3):497–505 doi:10.1039/C5LC01139F |
| 39 |
Villar G, Graham A D, Bayley H. Science, 2013, 340 (6128):48–52 doi:10.1126/science.1229495 |
| 40 |
Geng T, Novak R, Mathies R A. Anal. Chem., 2014, 86 (1):703–712 doi:10.1021/ac403137h |
| 41 |
Shih S C, Gach P C, Sustarich J, Simmons B A, Adams P D, Singh S, Singh A K. Lab Chip, 2015, 15 (1):225–236 doi:10.1039/C4LC00794H |
| 42 |
Huang L, Chen Y, Chen Y, Wu H. Anal. Chem., 2015, 87 (24):12169–12176 doi:10.1021/acs.analchem.5b03031 |
| 43 |
Li L, Wang Q, Feng J, Tong L, Tang B. Anal. Chem., 2014, 86 (10):5101–5107 doi:10.1021/ac500881p |
| 44 |
Liu X, Barizuddin S, Shin W, Mathai C J, Gangopadhyay S, Gillis K D. Anal. Chem., 2011, 83 (7):2445–2451 doi:10.1021/ac1033616 |
| 45 |
Burger R, Kurzbuch D, Gorkin R, Kijanka G, Glynn M, McDonagh C, Ducree J. Lab Chip, 2015, 15 (2):378–381 doi:10.1039/C4LC01002G |
| 46 |
Hegde M, Jindal R, Bhushan A, Bale S S, McCarty W J, Golberg I, Usta O B, Yarmush M L. Lab Chip, 2014, 14 (12):2033–2039 doi:10.1039/C4LC00071D |
| 47 |
Ruppen J, Wildhaber F D, Strub C, Hall S R, Schmid R A, Geiser T, Guenat O T. Lab Chip, 2015, 15 (14):3076–3085 doi:10.1039/C5LC00454C |
| 48 |
Matharu Z, Patel D, Gao Y, Haque A, Zhou Q, Revzin A. Anal. Chem., 2014, 86 (17):8865–8872 doi:10.1021/ac502383e |
| 49 |
Anene-Nzelu C G, Peh K Y, Fraiszudeen A, Kuan Y H, Ng S H, Toh Y C, Leo H L, Yu H. Lab Chip, 2013, 13 (20):4124–4133 doi:10.1039/c3lc50730k |
| 50 |
Shin C S, Kwak B, Han B, Park K. Mol. Pharm., 2013, 10 (6):2167–2175 doi:10.1021/mp300595a |
| 51 |
Kuo C T, Chiang C L, Chang C H, Liu H K, Huang G S, Huang R Y, Lee H, Huang C S, Wo A M. Biomaterials, 2014, 35 (5):1562–1571 doi:10.1016/j.biomaterials.2013.11.008 |
| 52 |
Niu Y, Bai J, Kamm R D, Wang Y, Wang C. Mol. Pharm., 2014, 11 (7):2022–2029 doi:10.1021/mp500054h |
| 53 |
Theberge A B, Yu J, Young E W, Ricke W A, Bushman W, Beebe D J. Anal. Chem., 2015, 87 (6):3239–3246 doi:10.1021/ac503700f |
| 54 |
Zhang J, Wu J, Li H, Chen Q, Lin J M. Biosens. Bioelectron., 2015, 68 :322–328 doi:10.1016/j.bios.2015.01.013 |
| 55 |
van der Meer A D, Orlova V V, ten Dijke P, van den Berg A. Mummery C L. Lab Chip, 2013, 13 (18):3562–3568 doi:10.1039/c3lc50435b |
| 56 |
Dereli-Korkut Z, Akaydin H D, Ahmed A H, Jiang X, Wang S. Anal. Chem., 2014, 86 (6):2997–3004 doi:10.1021/ac403899j |
| 57 |
Kim S, Lee H, Chung M, Jeon N L. Lab Chip, 2013, 13 (8):1489–1500 doi:10.1039/c3lc41320a |
| 58 |
Chen M B, Srigunapalan S, Wheeler A R, Simmons C A. Lab Chip, 2013, 13 (13):2591–2598 doi:10.1039/c3lc00051f |
| 59 |
Lee H, Kim S, Chung M, Kim J H, Jeon N L. Microvasc. Res., 2014, 91 :90–98 doi:10.1016/j.mvr.2013.12.001 |
| 60 |
Jusoh N, Oh S, Kim S, Kim J, Jeon N L. Lab Chip, 2015, 15 (20):3984–3988 doi:10.1039/C5LC00698H |
| 61 |
Grosberg A, Alford P W, McCain M L, Parker K K. Lab Chip, 2011, 11 (24):4165–4173 doi:10.1039/c1lc20557a |
| 62 |
Abaci H E, Gledhill K, Guo Z, Christiano A M, Shuler M L. Lab Chip, 2015, 15 (3):882–888 doi:10.1039/C4LC00999A |
| 63 |
Wagner I, Materne E M, Brincker S, Sussbier U, Fradrich C, Busek M, Sonntag F, Sakharov D A, Trushkin E V, Tonevitsky A G, Lauster R, Marx U. Lab Chip, 2013, 13 (18):3538–3547 doi:10.1039/c3lc50234a |
| 64 |
Huh D, Matthews B D, Mammoto A, Montoya-Zavala M, Hsin H Y, Ingber D E. Science, 2010, 328 (5986):1662–1668 doi:10.1126/science.1188302 |
| 65 |
Park J, Lee B K, Jeong G S, Hyun J K, Lee C J, Lee S H. Lab Chip, 2015, 15 (1):141–150 doi:10.1039/C4LC00962B |
| 66 |
Cho H, Seo J H, Wong K H, Terasaki Y, Park J, Bong K, Arai K, Lo E H, Irimia D. Sci. Rep., 2015, 5 :15222 doi:10.1038/srep15222 |
| 67 |
Choi Y, Hyun E, Seo J, Blundell C, Kim H C, Lee E, Lee S H, Moon A, Moon W K, Huh D. Lab Chip, 2015, 15 (16):3350–3357 doi:10.1039/C5LC00514K |
| 68 |
Jeon J S, Bersini S, Gilardi M, Dubini G, Charest J L, Moretti M, Kamm R D. P. Natl. Acad. Sci. USA, 2015, 112 (7):E818–E818 doi:10.1073/pnas.1501426112 |
| 69 |
Ruiz A, Joshi P, Mastrangelo R, Francolini M, Verderio C, Matteoli M. Lab Chip, 2014, 14 (15):2860–2866 doi:10.1039/c4lc00174e |
| 70 |
Rodriguez-Rodriguez R, Munoz-Berbel X, Demming S, Buttgenbach S, Herrera M D, Llobera A. Biomed. Microdevices, 2012, 14 (6):1129–1140 doi:10.1007/s10544-012-9679-y |
| 71 |
Zhang Y, Zhang W, Qin L. Angew. Chem. Int. Ed. Engl., 2014, 53 (9):2344–2348 doi:10.1002/anie.201309885 |
| 72 |
Kim C, Kasuya J, Jeon J, Chung S, Kamm R D. Lab Chip, 2015, 15 (1):301–310 doi:10.1039/C4LC00866A |
| 73 |
Espulgar W, Aoki W, Ikeuchi T, Mita D, Saito M, Lee J K, Tamiya E. Lab Chip, 2015, 15 (17):3572–3580 doi:10.1039/C5LC00652J |
| 74 |
Li L, Wang W, Ding M, Luo G, Liang Q. Anal. Chem., 2016, 88 (13):6734–6742 doi:10.1021/acs.analchem.6b01008 |
| 75 |
Du G S, Pan J Z, Zhao S P, Zhu Y, den Toonder J M, Fang Q. Anal. Chem., 2013, 85 (14):6740–6747 doi:10.1021/ac400688f |
| 76 |
Ding Y, Li J, Xiao W, Xiao K, Lee J, Bhardwaj U, Zhu Z, Digiglio P, Yang G, Lam K S, Pan T. Anal. Chem., 2015, 87 (20):10166–10171 doi:10.1021/acs.analchem.5b00826 |
| 77 |
Lou X, Kim G, Yoon H K, Lee Y E, Kopelman R, Yoon E. Lab Chip, 2014, 14 (5):892–901 doi:10.1039/c3lc51077h |
| 78 |
An D, Kim K, Kim J. Biomol. Ther. (Seoul), 2014, 22 (4):355–362 doi:10.4062/biomolther.2014.078 |
| 79 |
Chen S Y, Hung P J, Lee P J. Biomed. Microdevices, 2011, 13 (4):753–758 doi:10.1007/s10544-011-9545-3 |
| 80 |
Wu Q, Gao D, Wei J, Jin F, Xie W, Jiang Y, Liu H. Chem. Commun. (Camb), 2014, 50 (21):2762–2764 doi:10.1039/c3cc49771b |
| 81 |
Gao D, Li H, Wang N, Lin J M. Anal. Chem., 2012, 84 (21):9230–9237 |
| 82 |
Qiao W, Wang C, Ding Z, Song J, Wei X, Lo Y H. Microfluid. Nanofluid., 2016, 20 (5):77 doi:10.1007/s10404-016-1743-0 |
| 83 |
Reedy C R, Price C W, Sniegowski J, Ferrance J P, Begley M, Landers J P. Lab Chip, 2011, 11 (9):1603–1611 doi:10.1039/c0lc00597e |
| 84 |
Abate A R, Hung T, Sperling R A, Mary P, Rotem A, Agresti J J, Weiner M A, Weitz D A. Lab Chip, 2013, 13 (24):4864–4869 doi:10.1039/c3lc50905b |
| 85 |
Pinheiro L B, Coleman V A, Hindson C M, Herrmann J, Hindson B J, Bhat S, Emslie K R. Anal. Chem., 2012, 84 (2):1003–1011 doi:10.1021/ac202578x |
| 86 |
Fu Y, Li C, Lu S, Zhou W, Tang F, Xie X S, Huang Y. Proc. Natl. Acad. Sci. USA, 2015, 112 (38):11923–11928 doi:10.1073/pnas.1513988112 |
| 87 |
Estes M D, Yang J, Duane B, Smith S, Brooks C, Nordquist A, Zenhausern F. Analyst, 2012, 137 (23):5510–5519 doi:10.1039/c2an35768b |
| 88 |
ZHAO Shu-Mi, ZHU Ling, ZHU Can-Can, LI Yang, WANG Hua-Dong, ZHANG Long, DU Di-Wei, DENG Guo-Qing, WANG An, LIU Yong. Chinese J. Anal. Chem., 2014, 42 (10):1393–1399 doi:10.1016/S1872-2040(14)60770-2 赵树弥, 朱灵, 朱灿灿, 李阳, 王华东, 张龙, 堵棣威, 邓国庆, 王安, 刘勇. 分析化学, 2014 ,42(10) :1393–1399 doi:10.1016/S1872-2040(14)60770-2 |
 2016, Vol. 44
2016, Vol. 44